
생화학분자생물학회입니다.
TALEN 유전자 가위를 이용한 Rh D 혈액형의 변환
작성자
관리자작성일자
2015-12-01조회수
532TALEN 유전자 가위를 이용한 Rh D 혈액형의 변환
Rh D Blood Group Conversion Using Transcription Activator-Like Effector Nucleases Nat. Commun. 6: 7451, 2015

이용석 |

김영훈 |

김형범 |
연구배경
Zinc-Finger Nucleases(ZFNs), Transcription Activator-Like Effector Nucleases(TALENs) and RNA-Guided Engineered Nucleases(RGENs) 등의 유전자가위는 세포와 생물체에서 유전자 교정/편집을 수행해 낸다 (7). 유전자가위를 이용한 유전자 조작은 유전자 파괴, 삽입, 교정, 유전자 재배열등을 가능하게 해준다. 유전자파괴는 유전자가위를 이용한 가장 간단한 형태의 유전자 조작이며, 이는 하나 혹은 한 쌍의 유전자가위를 사용하여 Double-Stranded Break를 특정 부위에 일으킬 수 있게 한다. 유전자가위로 유도된 Double-Stranded Breaks 는 Error-Prone Non Homologous-End Joining 을 통해 복원될 수 있으며, 이때 생기는 Indel(Insertion and Deletion)이 유전자 파괴를 유도하게 된다.
우리 팀은 유전자가위를 사용하여 혈액형을 결정하는 유전자를 조작하는 것이 혈액형 전환으로 이어질 수 있다고 가정하고 Rh D+형의 인간 적혈구 전구세포에 2가지 다른 쌍의 TALENs를 이용하여 RHD 유전자를 파괴했다. 이를 통해 RHD-Knockout 적혈구 전구세포를 얻었고 유세포 분석기와 응집반응검사를 통해 Rh D- 표현형을 보이는 것을 확인할 수 있었다. 이 결과는 유전자가위를 이용한 유전자 조작으로 혈액형 전환이 가능하다는 것을 증명해 준다.
연구결과
RHD 유전자를 목표로 하는 TALENs의 디자인과 검증
확실하게 RHD 유전자 발현을 막기 위해 우리 팀은 우선 단백질 암호화 부분(Coding Region)을 타겟팅하는 TALEN쌍을 준비했다. NCBI와 Ensembl에 있는 전사 정보에 따르면 인간 RHD 유전자는 공통적으로 10가지 Transcript Variants를 가지고 있으며 첫 번째 엑손은 모든 Transcript Variants에 포함되기 때문에 첫 번째 엑손을 목표로 하는 TALEN 쌍을 우선 준비했다 (RHD_E1_TALENs; 그림. 1a). 게다가 네 번째 엑손은 일부 Rh D- 사람들에게 유전자 돌연변이가 생기는 부분이다. 따라서 우리 팀은 네 번째 엑손을 목표로 하는 TALEN 또한 디자인하였다 (RHD_E4_TALENs; 그림 1a).
디자인한 TALENs의 작용여부를 확인하기 위하여 HEK293T세포에 플라스미드를 Transfection 시키고 돌연변이율을 확인할 수 있는 T7E1 를 시행하였다. RHD의 첫 번째와 네 번째 엑손의 목표로 한 부분에서의 변이 빈도는 각각 12%, 6% 였으며 (그림. 1b, c) 이는 두 쌍의 TALENs 모두 활성이 있음을 알려준다.
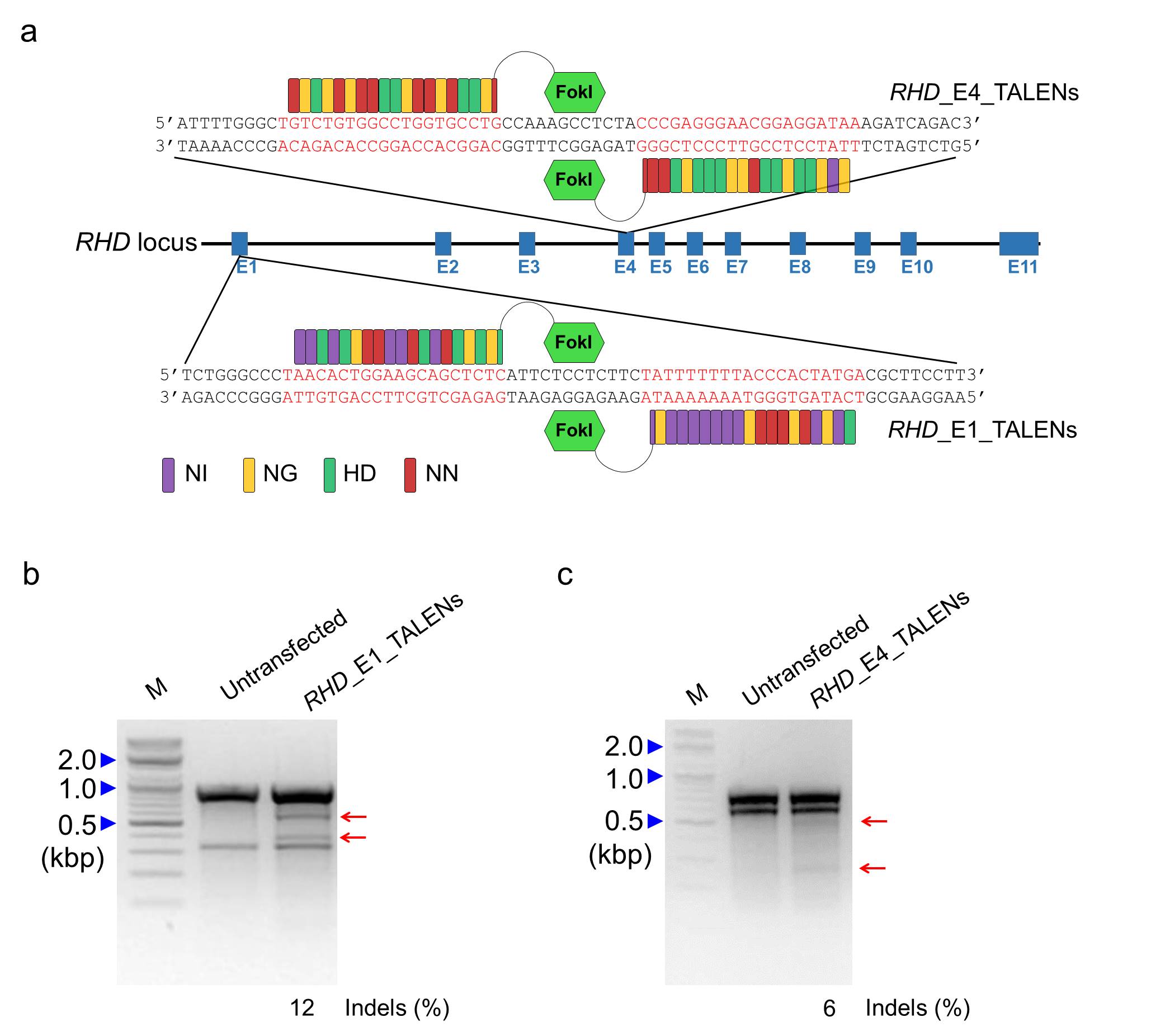
인간 RHD 유전자 표적 TALEN (a) RHD 유전자의 첫 번째 엑손(E1)과 네 번째 엑손(E4) 목표로 하는 TALEN (b) T7E1 Assay 를 이용한 TALEN의 활성도 검사. 그림은 Nat. Commun. 6: 7451, 2015 에 발표되었음.
RHD 유전자 변이를 가진 클론 생산
우리 팀은 위에서 만든 TALENs를 이용하여 Rh D+형 적혈구 전구세포에서 RHD-Knockout 세포를 만드는 시도를 했다. 적혈구 전구세포로는 Rh D+형(DD) 기부자의 섬유아세포에서 만들어진 유도만능줄기세포에서 유도된 HiDEP-1 세포주를 사용했다 (8). HiDEP-1세포에 RHD_E1_TALENs 또는 RHD_E4_TALENs를 발현하는 플라스미드를 HiDEP-1 세포에 Transfection 시키고 클론을 만들기 위해 96well 판에 0.25cell/well이 되도록 심었다. 이후 얻어진 클론들은 모두 T7E1 Assay 를 통하여 돌연변이 여부를 1차적으로 검증하였다. 결과 RHD_E1_TALENs를 이용하여 첫 번째 엑손을 목표로 한 경우 9개의 클론에서 돌연변이를 발견할 수 있었고 RHD_E4_TALENs를 이용하여 네 번째 엑손을 목표로 한 경우 7개의 클론에서 돌연변이를 발견할 수 있었다 (그림. 2). 이후 시퀀싱을 통해 각각 선별된 돌연변이 클론의 RHD 유전자를 확인하였다. 그 결과 첫 번째 엑손을 타겟팅하는 TALEN을 사용한 경우 Biallelic Out of Frame 돌연변이를 가지고 있던 한 클론을 분리하여 E1_B로 명명하고 추후 진행되는 실험에 사용하였다. 마찬가지로 네 번째 엑손을 목표로 한 경우 Biallelic Out of Frame 돌연변이를 가지는 한 클론과 Monoallelic out of frame 돌연변이를 가지는 한 클론을 선택하여 E4_B 와 E4_M으로 명명하고 추후 실험에 사용하였다.
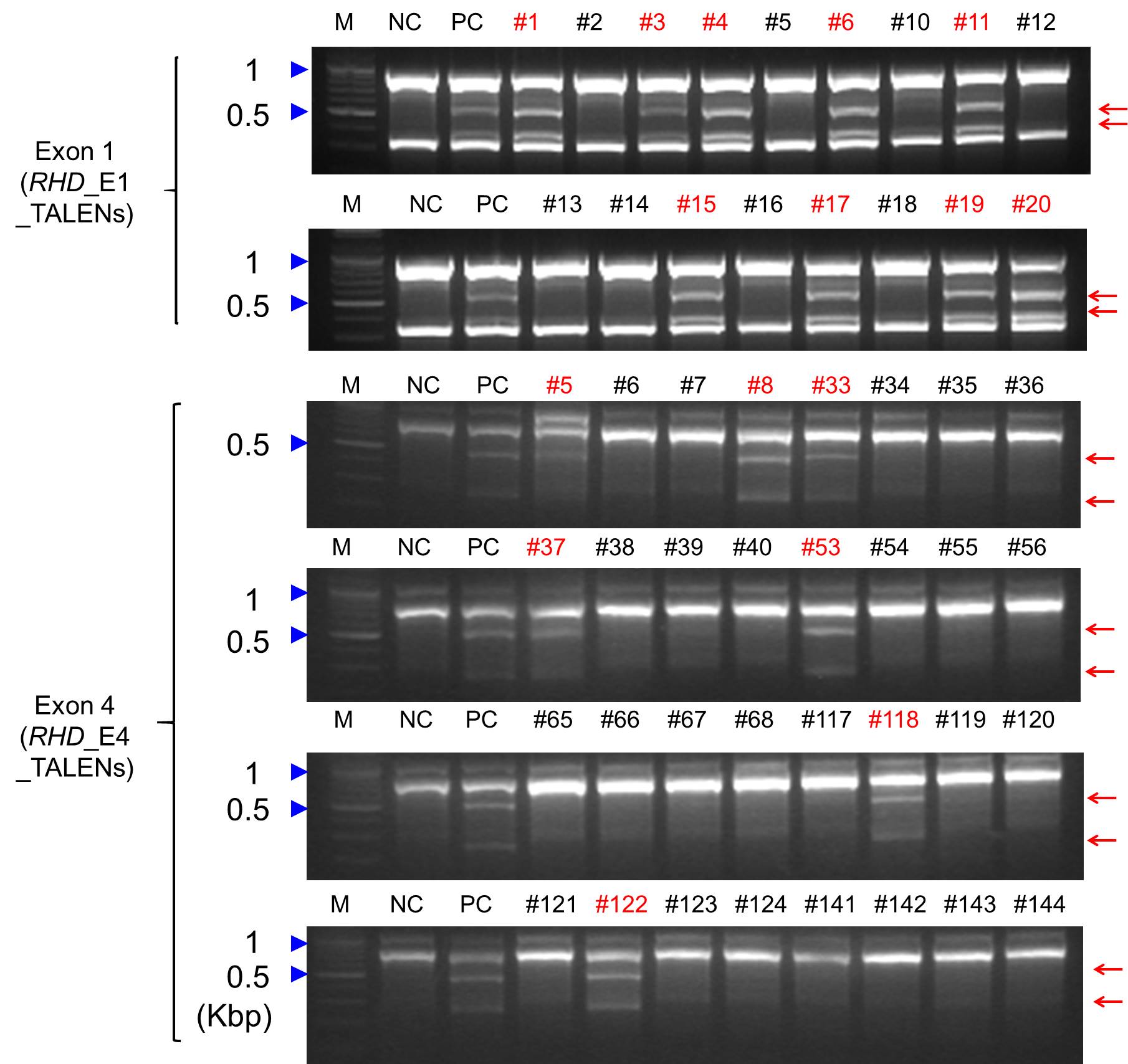
그림. 2
RHD 유전자가 변이된 적혈구 전구세포주 구축을 위한 클론 분석. 클론 분석은 T7E1 Assay 를 통해 이루어짐. 변이가 일어난 클론은 빨강색 번호로 표시함. 그림은 Nat. Commun. 6: 7451, 2015 에 발표되었음.
그 후 우리 연구팀은 다음 실험에 사용할 돌연변이세포들이 RHD 엑손 영역에 PTC(Premature Termination Codon)를 가지는지 확인하기 위해 시퀀싱 결과를 검토했다. E1_B의 두 대립유전자 모두 첫 번째 엑손에 PTC를 가지고 있었다 (그림. 3a). E4_B의 경우 한 쪽은 네 번째 엑손에 다른 한 쪽은 다섯 번째 엑손에 PTC를 가지고 있었다 (그림. 3b). 이 정보들을 종합하면 결과적으로 E4_M클론에서는 RHD 단백질이 발현될 것이나 E1_B와 E4_B에서는 발현이 되지 않을 것임을 알 수 있다.
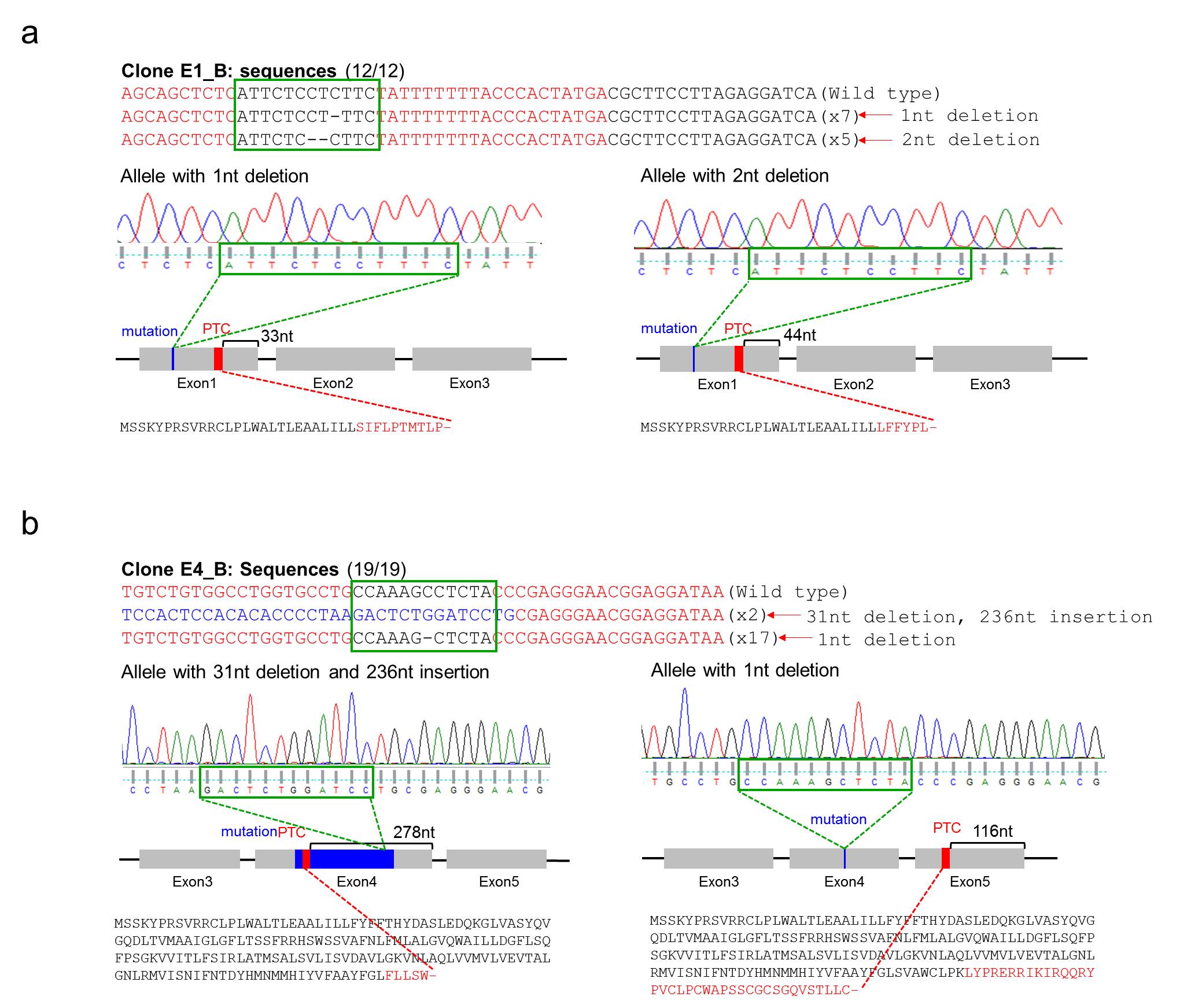
그림. 3
RHD 유전자가 변이된 클론의 DNA 시퀀싱 결과. (a) RHD 유전자의 첫 번째 엑손이 Biallelic 변이된 클론의 시퀀싱 결과. (b) RHD 유전자의 네 번째 엑손이 Biallelic 변이된 클론의 시퀀싱 결과. (c) RHD 유전자의 첫 번째 엑손이 Monoallelic 변이된 클론의 시퀀싱 결과. 그림은 Nat. Commun. 6: 7451, 2015 에 발표되었음.
RHD Knockout 클론에서 D항원의 결손
우리 팀은 이후에 유세포 분석기를 사용하여 각각의 HiDEP-1 세포에서 D항원단백질 표현 여부를 평가했다. 결과의 Positive Control과 Negative Control을 위해 각각 Rh D+와 Rh D- 기증자들로부터 분리된 혈액을 사용했다. 적혈구를 의미하는 표지단백질인 Glycophorin A를 발현하는 세포 중에서 D항원의 발현율은 Rh D+와 Rh D-에서 각각 99.96 ± 0.004%와 1.4% ± 0.6%였으며 (그림. 4), 이는 Flow Cytometry가 1% 정도의 작은 오류(Nonspecific Background Signal)로 D항원 발현 여부를 확인할 수 있는 민감한 방법이라는 것을 의미한다.
각 클론들은 4 일동안 적혈구로의 분화가 유도되었고 그 후 유세포분석기를 통해 분석하였다. Glycophorin A+ 세포들 중 Parental 클론과 Monoallelic 돌연변이 클론들은 현저한 D 항원 표현율을 보였다 (그림. 4; Parental 클론: 93±0.9%, Monoallelic 돌연변이 클론: 73±2%). 하지만 Monoallelic 돌연변이 클론의 D항원 발현율은 Parental 클론의 D항원 표현율에 비해 크게 낮았는데 (P<0.001, ANOVA), 이는 Monoallelic 돌연변이가 D항원의 발현율을 낮춘다는 것을 의미한다. 모든 RHD Biallelic 돌연변이 클론에서 D항원 발현율은 Nonspecific Background Signal 수치와 유사했고 이는 TALEN으로 유도한 RHD Biallelic 돌연변이 클론에서 D항원 발현이 되지 않고 있다는 것을 의미한다.
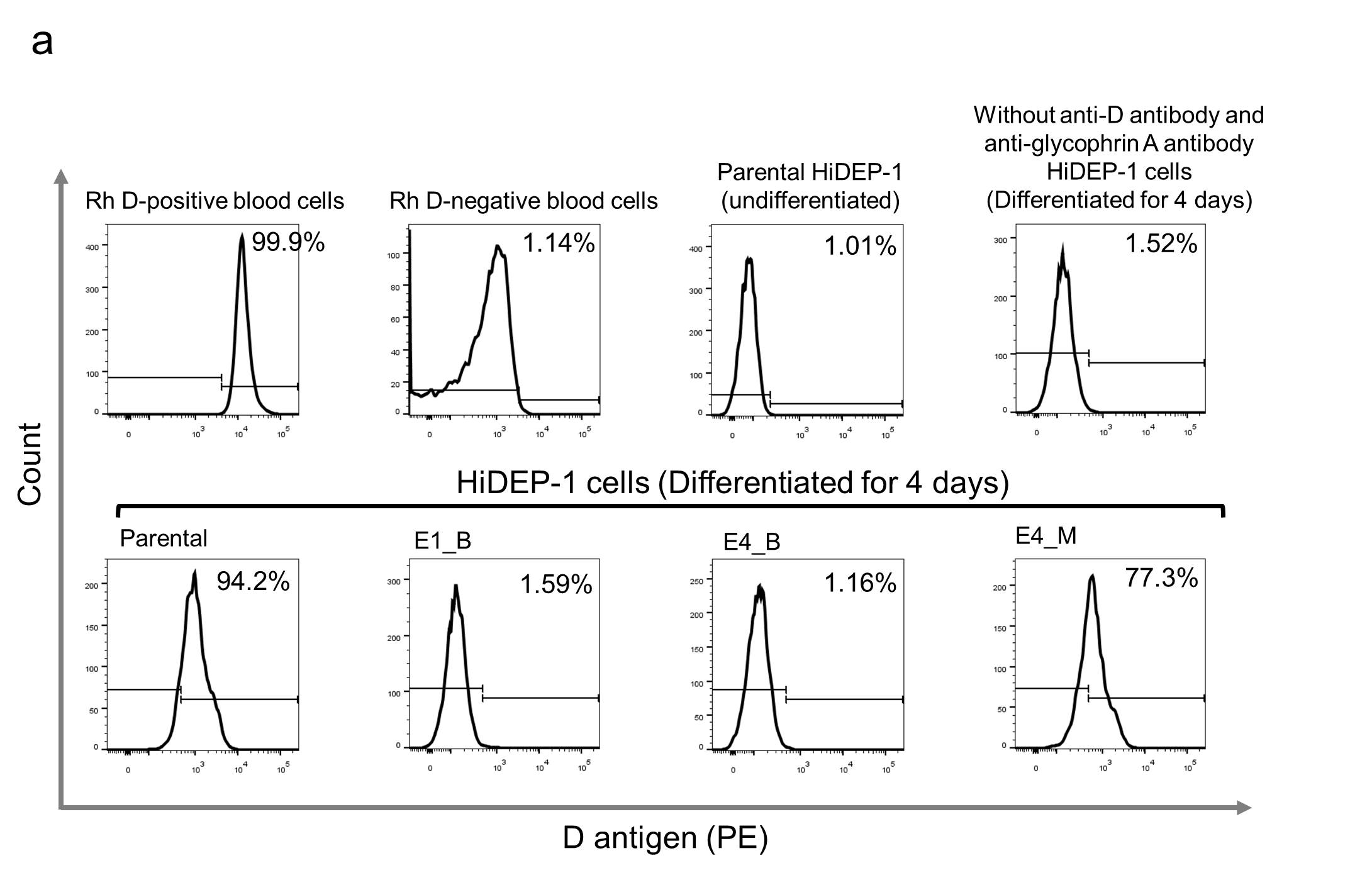
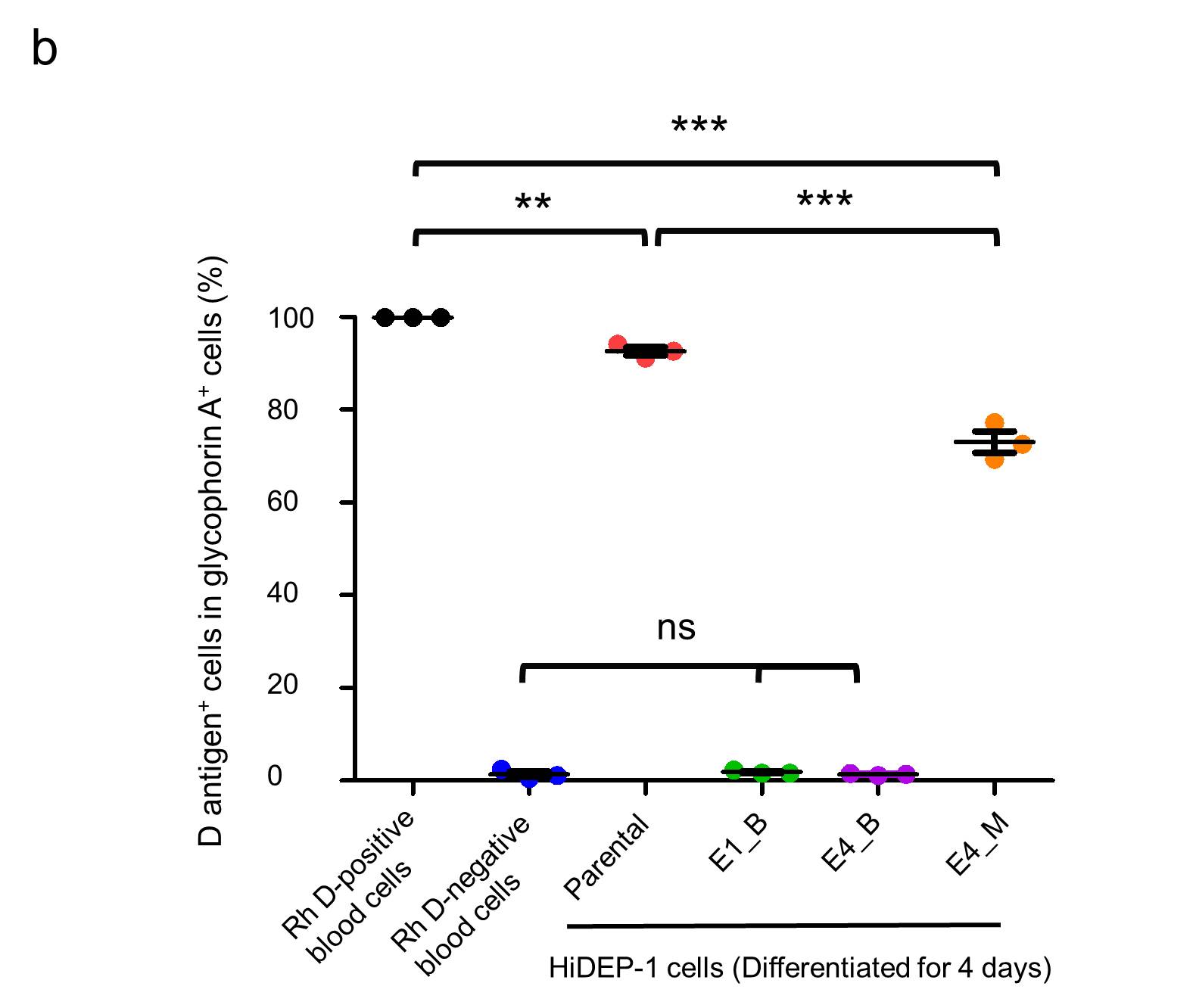
그림. 4
유세포 분석기를 이용한 RHD Knockout 클론의 D항원 발현 조사. Parental 클론과 RHD 유전자가 변이된 클론을 4 일동안 분화 후 유세포 분석기를 통해 분석함. (a) 대표적인 유세포 분석. (b) glycophorin A를 발현하는 세포 중 D항원의 발현율. **P < 0.01, ***P < 0.001. 그림은 Nat. Commun. 6: 7451, 2015 에 발표되었음.
Knockout 클론의 응집반응검사
우리는 그 후 혈액형 검사에 쓰이는 응집반응검사을 시행했다. Parental, RHD Knockout (E1_B, E4_B) 그리고 RHD Monoallelic 돌연변이 (E4_M) HiDEP-1 세포들을 4일 동안 분화를 유도시킨 후 Rh D형 검증에 쓰이는 응집반응검사를 시행하였다. Positive Control과 Negative Control을 위해 각각 응집과 비응집이라는 결과를 보이는 Rh D+형과 Rh D-형의 혈액을 사용했다. Parental과 E4_M의 경우 응집이 일어났고 E1_B와 E4_B는 그렇지 않았는데 (그림. 5a), 이는 E1_B와 E4_B의 표현형적으로 Rh D-라는 것을 의미한다.
위에서 사용된 평범한 응집반응검사로는 희귀 혈액형 중 하나인 Weak D 혈액형 역시 비응집으로 나타날 수 있다. Weak Rh D+ 세포 존재 여부를 확인하기 위해 Weak D 응집반응검사를 시행했다. 이 실험에서 두 클론 모두 응집반응이 일어나지 않았으며 (그림. 5b), 이는 이 두 RHD Knockout 클론들이 약한 Rh D+형이 아니라 Rh D-형이라는 것을 증명해준다.
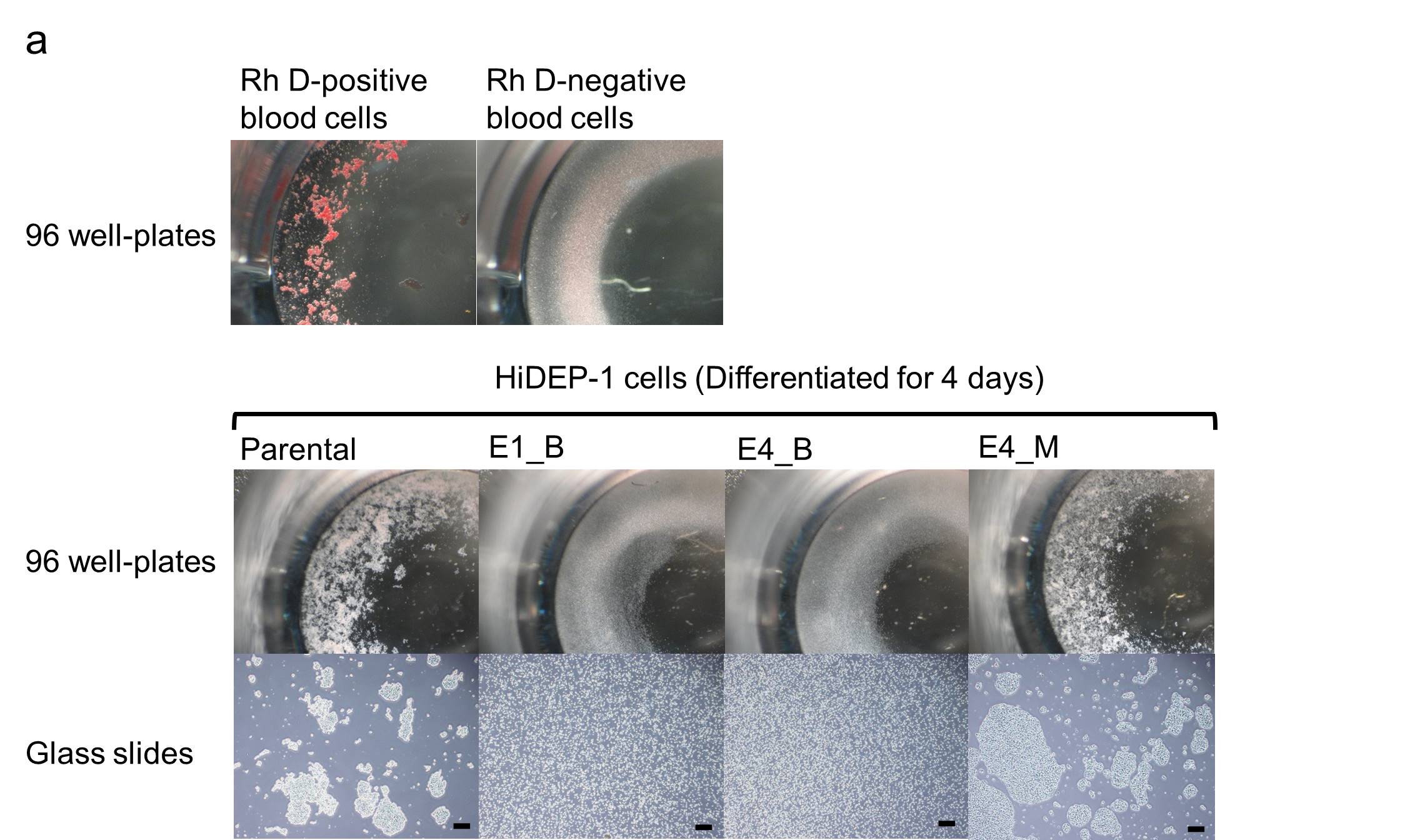
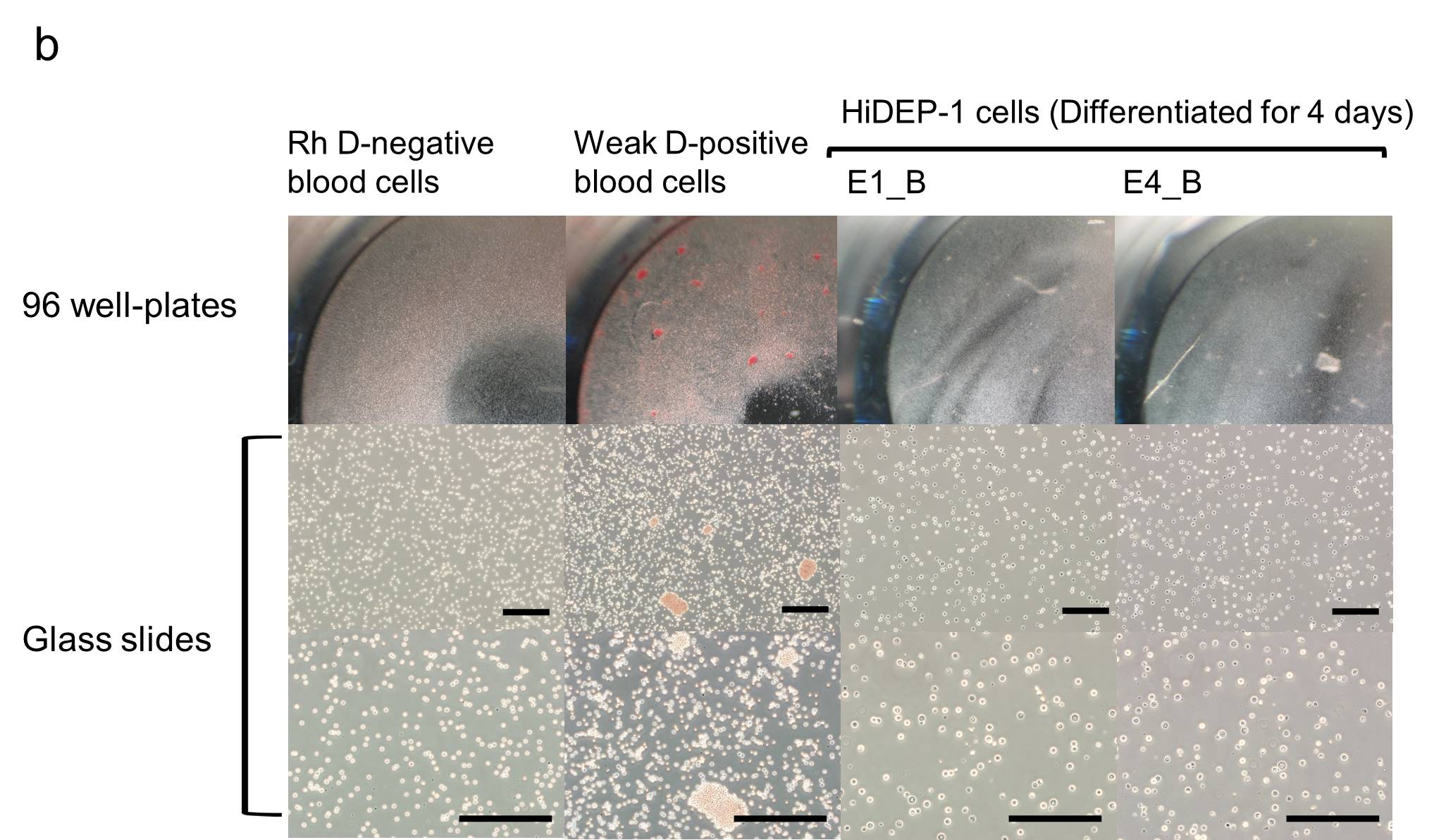
그림. 5
RHD Knockout 클론의 응집반응 검사 (a) Parental 클론과 RHD 유전자가 변이된 클론은 4일 동안 분화 후 응집반응 검사를 진행함. (b) Weak D 혈액을 이용한 Parental 클론과 RHD 유전자가 변이된 클론의 Weak D 응집반응검사. 그림은 Nat. Commun. 6: 7451, 2015 에 발표되었음. Scale = 500 mm.
RHD Knockout 클론의 분화와 작용
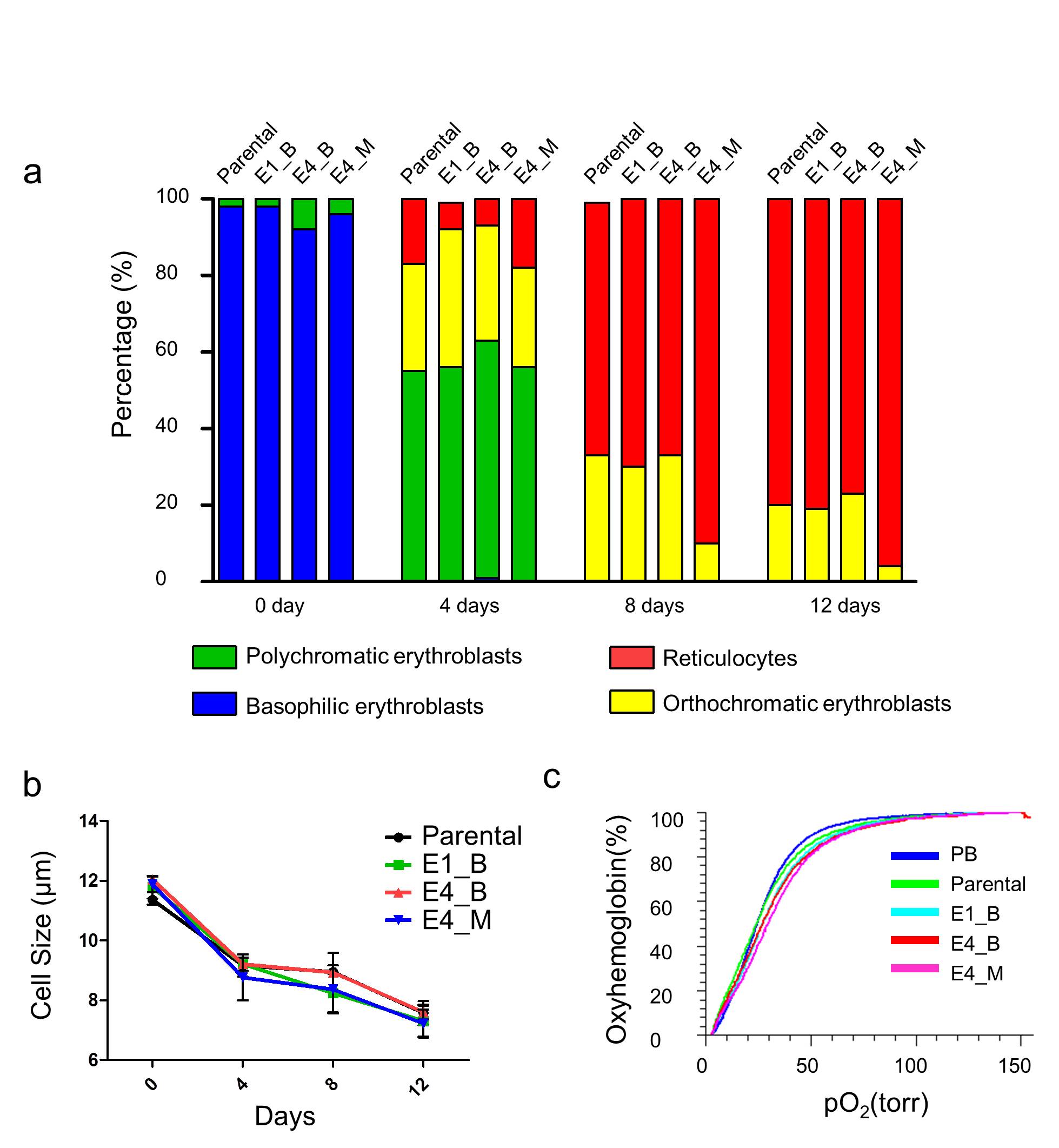
그림. 6
RHD Knockout 클론의 분화와 기능 평가. (a) 모든 클론은 적혈구 분화를 진행하여 Wright-Giemsa 염색을 통해 분화 정도 분석. (b) 적혈구 분화 동안 모든 클론의 평균 세포크기 분석 (c) 모든 클론의 산소 결합 및 해리 곡선 분석을 통한 산소 전달 능력 분석. 그림은 Nat. Commun. 6: 7451, 2015 에 발표되었음.
연구의 성과 및 의의
Rh D-형은 혈액형이 맞지 않거나 혈액형이 미리 확인될 수 없는 응급상황에서도 수혈될 수 있는 만능혈액이기 때문에 다른 혈액형들을 O Rh D-형으로 전환하는 방법에 관해 수혈의학에서 오랫동안 연구되어 왔다. ABO 혈액형은 Glycosyltransferase의 활동으로 정해지기 때문에 A 또는 B형을 O형으로 전환하는 것은 효소반응을 이용하여 성취될 수 있었다 (9). 그러나 RHD형은 특정 효소의 작용 여부가 아니라 RHD 항원 단백질의 발현 여부로 정해지기 때문에 효소반응을 이용한 유사한 방법들은 적용이 사실상 불가능하다. 여기서 우리 팀은 TALEN을 기반으로 RHD를 파괴하여 Rh D+ 세포를 Rh D- 세포로 전환할 수 있다는 것을 보였다.
이전 연구들에서 효소를 이용해 다른 혈액형을 O형으로 전환하는 방법과 비교하여 우리의 접근 방식은 장점과 단점이 있다. 우리의 접근 방식은 적혈구 전구세포나 줄기세포에 적용되어 수많은 적혈구들을 생산해 낼 수 있다. In vivo 또는 ex vivo에서 조혈모세포에 유전자가위를 적용한다면 전환된 혈액형을 가지는 적혈구들을 거의 무제한적으로 생산할 수 있게 될 것이다. 그러나 이러한 유전자 조작법은 효소를 이용한 방법과는 다르게 이미 성숙하여 탈핵이 완료된 적혈구에는 적용이 될 수 없다. 두 접근 방식에서 혈액형 전환의 방법이 다르다는 것을 볼 때 두 방법이 혼합되어 새로운 효율적인 혈액형 전환법이 개발될 가능성도 있다.
다른 종류의 유전자가위들이 유전자 수술 능력과 메커니즘을 공유하는 것에 착안하면, Rh D-형으로의 혈액형 전환이 ZFNs와 RGENs를 이용해서도 성취될 수 있다고 생각할 수 있다. 나아가 우리의 연구는 유전자가위가 다른 Rh D형이 아닌 다른 혈액형의 전환에도 사용될 수 있는 가능성을 제시한다. 예를 들어, ABO형은 ABO유전자로 정해지기 때문에 유전자가위를 사용한 ABO 유전자의 파괴는 A, B 또는 AB형의 세포로부터 O형으로의 전환을 이끌어 낼 수 있다. 유전자가위를 이용한 적혈구 전구세포 또는 조혈모세포에서의 ABO와 RHD 동시파괴는 만능혈액세포인 Rh D- O형 혈액의 생산을 가능하게 할 것이다.
요약하자면 우리는 본 연구에서 유전자가위의 적용 범위를 혈액형 전환에까지 확장시켰으며, TALENs를 이용해 RHD 유전자파괴를 유도함으로써 Rh D+ 세포를 Rh D- 세포로 전환할 수 있다는 것을 보였다. 이 유전자가위를 기반으로 하여 유전자 조작을 통해 혈액형을 바꾸는 기법은 수혈의학에 있어 혈액형 전환 방법의 개발에 새로운 길을 열어준다.
참고문헌
| 1. | Landsteiner, K. WA. (1940) An agglutinable factor in human blood recognized by immune sera for rhesus blood. Proc Soc Exp Biol Med 43, 223. |
| 2. | Daniels, G. (2009) The molecular genetics of blood group polymorphism. Hum Genet 126, 729-742. |
| 3. | Le van Kim, C., Mouro, I., Cherif-Zahar, B. et al. (1992) Molecular cloning and primary structure of the human blood group RhD polypeptide. Proc Natl Acad Sci U S A89, 10925-10929. |
| 4. | Cherif-Zahar, B., Mattei, MG., Le Van Kim, C., Bailly, P., Cartron, JP. and Colin, Y. (1991) Localization of the human Rh blood group gene structure to chromosome region 1p34.3-1p36.1 by in situ hybridization. Hum Genet 86, 398-400. |
| 5. | Daniels, G. (2013) Human blood groups : Geoff Daniels ; foreword to first edition by Ruth Sanger, 3rd ed, John Wiley & Sons, Chichester, West Sussex. |
| 6. | Daniels, G. (2013) Variants of RhD--current testing and clinical consequences. Br J Haematol 161, 461-470. |
| 7. | Kim, H. and Kim, JS. (2014) A guide to genome engineering with programmable nucleases. Nat Rev Genet 15, 321-334. |
| 8. | Kurita, R., Suda, N., Sudo, K. et al. (2013) Establishment of immortalized human erythroid progenitor cell lines able to produce enucleated red blood cells. PLoS One 8, e59890. |
| 9. | Liu, QP., Sulzenbacher, G., Yuan, H. et al. (2007) Bacterial glycosidases for the production of universal red blood cells. Nat Biotechnol 25, 454-464. |
첨부파일