
생화학분자생물학회입니다.
Chemical Biology Laboratory (분자인식연구센터 화학생물학연구실)
작성자
관리자작성일자
2017-04-01조회수
425Chemical Biology Laboratory (분자인식연구센터 화학생물학연구실)
이준석
한국과학기술연구원, UST-KIST Campus 생명화학과
[연구실소개]
본 실험실에서는 다양한 생명현상을 이해하는데 이용할 수 있는 저분자툴(Small Molecule Toolbox)을 개발하고 이를 활용하여 병원균과 숙주세포간의 상호작용에 대한 연구를 한다. 화학생물학이라는 분야는 유기화학, 생화학 및 세포 분자생물학을 융합하는 학문으로서, 화학자의 눈과 새로운 화학적 분석기법의 개발을 통해 기존에 관찰하기 어렵고 풀기 어려웠던 생명현상 신비를 새로운 시각으로 접근하여 연구하는 학문이다. 우리 연구실에서는 감염질환에 대한 메커니즘 연구에 있어서 (1) 새로운 저분자 프로브를 활용하여 기존의 항체기반 어세이들을 대체할 수 있는 고감도의 형광프로브기반 어세이의 개발, (2) 살아있는 세포에서 실시간으로 단백질의 이동과 이들의 상호작용을 영상화 하는 기법의 개발, (3) 효소활성기반 프로테오믹스 연구가 활발히 진행 중이다.
[연구내용]
1. Proximity-Based Photo-Crosslinking Probe
전통적으로 단백질-단백질 상호작용(Protein-Protein Interaction, PPI)은 효모단백질잡종법(Yeast Two-Hybrid, Y2H) 혹은 항체기반 면역침강법(Immunoprecipitation, IP)을 이용하여 상호간의 단백질 결합을 연구해 왔다. 하지만 이들 연구기법의 경우 살아있는 생물의 환경(Intact Environment)과는 매우 동떨어진 환경에서 측정되는 단백질 결합력에 의존할 수 밖에 없다. 본 연구실에서는 이같은 현재 기술의 한계를 극복하기 위한 전략으로 광가교결합 반응과 형광에 관심을 갖게 되었다. 살아있는 세포의 환경에서 PPI를 측정하기 위하여 시공간 특이적으로 화학결합을 유도할 수 있는 광가교결합 작용기를 형광체에 도입하는 전략을 취하여, 형광광가교결합프로브를 개발할 수 있었다. 이 프로브를 활용하면, 특정 단백질과 상호작용하는 파트너 단백질을 형광으로 표지하는 것이 가능하다. 그 한가지 예로서, 단백질 혼합물 상에서 Cul2 형의 ubiquitin E3 ligase 의 기질만을 특이적으로 표지하고 형광 시그널을 이용하여 영상화 하는 것이 가능함을 실증할 수 있었다. 이처럼 광가교결합프로브를 활용하면 살아있는 세포의 Intact 상태에서 단백질들의 다이나믹한 상호작용과정을 효과적으로 연구하는 것이 가능하다.
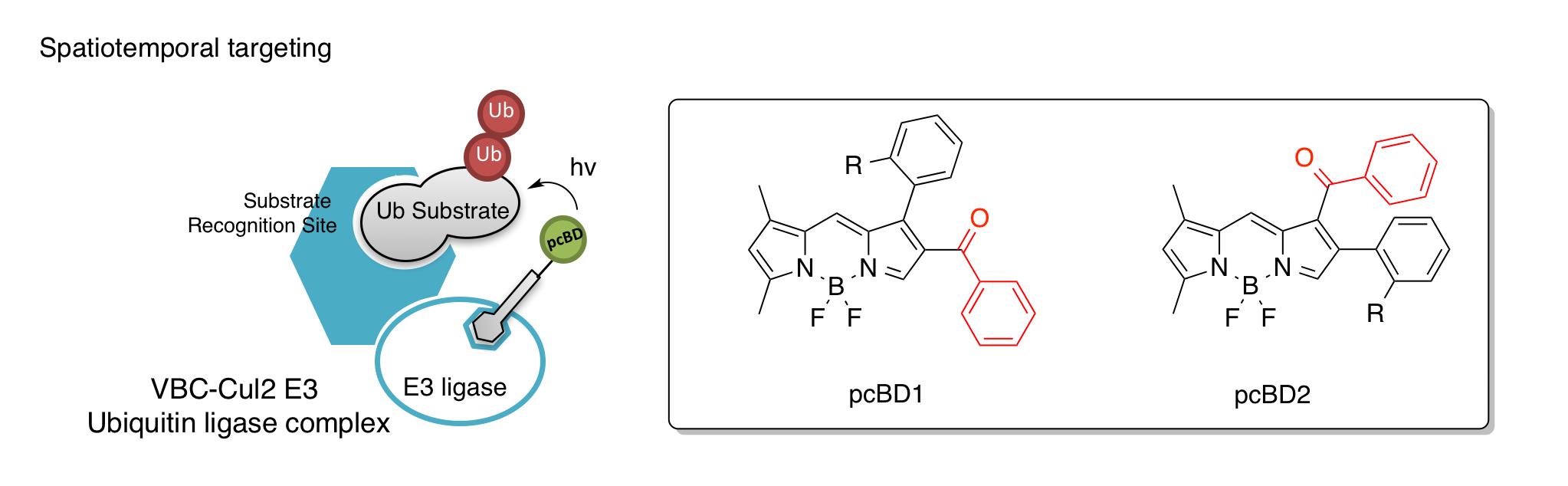
그림 1. 광가교결합형광프로브 (Photo-Crosslinking Fluorescence Probe Design) 구조 및 광가교결합 도식도
2. Chemical Proteomic Profiling for Host-Pathogen Interaction
Hepatitis C Virus(HCV) 감염은 전 세계적으로 발생하고 있으며 미국 내 간 이식 수술의 주 원인으로, 80 퍼센트 가량의 환자가 만성 질환을 앓고 있다. 지난 15 년 간, HCV 감염을 치료하기 위한 연구는 감염의 메커니즘이 명확하게 규명되지 않아, 치료법 개발이 매우 한정되어 있었다. 최근 여러 연구팀에서는 HCV 복제를 돕는 숙주(Host) 세포 내의 여러 인자(Factor)들에 주목하고 있다. 일반적인 효소는 구조단백질(Structural Protein)에만 관여하는 것으로 알려져 있는데, 바이러스성 효소들은 비구조적 단백질(Nonstructural Protein)도 잘라 HCV 증식에 도움을 주는 것이 밝혀졌다. 따라서 HCV 증식에 긴밀한 영향을 주는 숙주 세포 내의 인자들을 활용한다면 HCV 감염을 조절하는 치료제로써 큰 가능성을 가지게 될 것이라 기대되고 있다. 본 연구팀에서는 효소 활성 기반 프로파일링(Activity-Based Protein Profiling)의 보완적인 방법으로써, 전사체 발현기반 단백질 프로파일링 기법(Transcriptome Expression Assisted Non-Directed Proteomic Profiling, TEAnDPP)을 개발하였다. 이는 표적 단백질을 극도로 다양화 한다는 측면에서 큰 장점을 가지고 있는데, 특히 표적 단백질의 경향성을 부여하는 전략으로 다양한 친전자체(Electrophile)를 가지고 있는 화합물 프로브를 활용하였다. 각 작용기들은 시스테인 잔기(Cysteine Residue)와 결합하여 신호의 구별을 극대화 할 뿐 아니라 단백질 반응성에 따른 지문(Fingerprint)을 나타낼 것이라 예상하였고, 결과적으로 병원균 샘플에서 단백질 반응에 따른 패턴을 찾아낼 수 있었다.
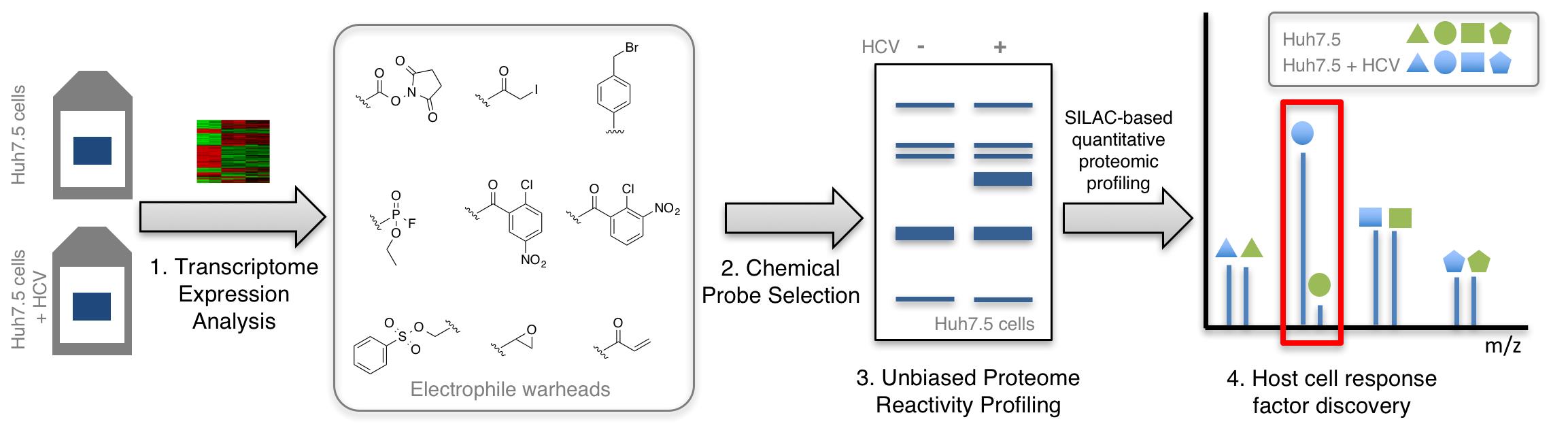
그림 2. 전사체 발현기반 단백질 프로파일링 기법(Transcriptome Expression Assisted Non-Directed Proteomic Profiling; TEAnDPP) 도식도
본 연구팀은 TEAnDPP 방법을 통해서 HCV 복제를 저해시키는 숙주세포 인자인 T-Plastin단백질을 찾아내었다. 이 실험결과를 통해 크게 두 가지 의미를 둘 수가 있는데, 첫 번째로 HTS 기반의 분석적 측면에서 적절한 화합물 프로브의 스크리닝에 도움을 줄 것이고, 두 번째로 HCV 연구에 있어서 T-Plastin의 선택적인 활성 저해가 HCV 감염 치료에 새로운 대안이 될 수 있을 것으로 기대한다.
3. Fluorescence Library for Biomolecule Sensors
분자영상(Molecular Imaging)은 살아있는 생명체에서 일어나는 다양한 분자수준의 생화학적 변화를 영상화하는 기법이다. 이때 이용하는 화합물을 분자영상 프로브(Molecular Imaging Probe)라 하는데, 이 중 분자영상에 이용하는 시그널이 형광(Fluorescence)인 화합물을 형광프로브 (Fluorescence Probe)라 명명한다. 형광프로브는 기능적 측면에서 크게 두 가지로 나눠 볼 수 있는데, 첫 번째는 이미징 표지자(Imaging Tag) 기능이고, 두 번째 형광 프로브의 기능으로는 화학센서(Chemosensor)로써 기능이다.
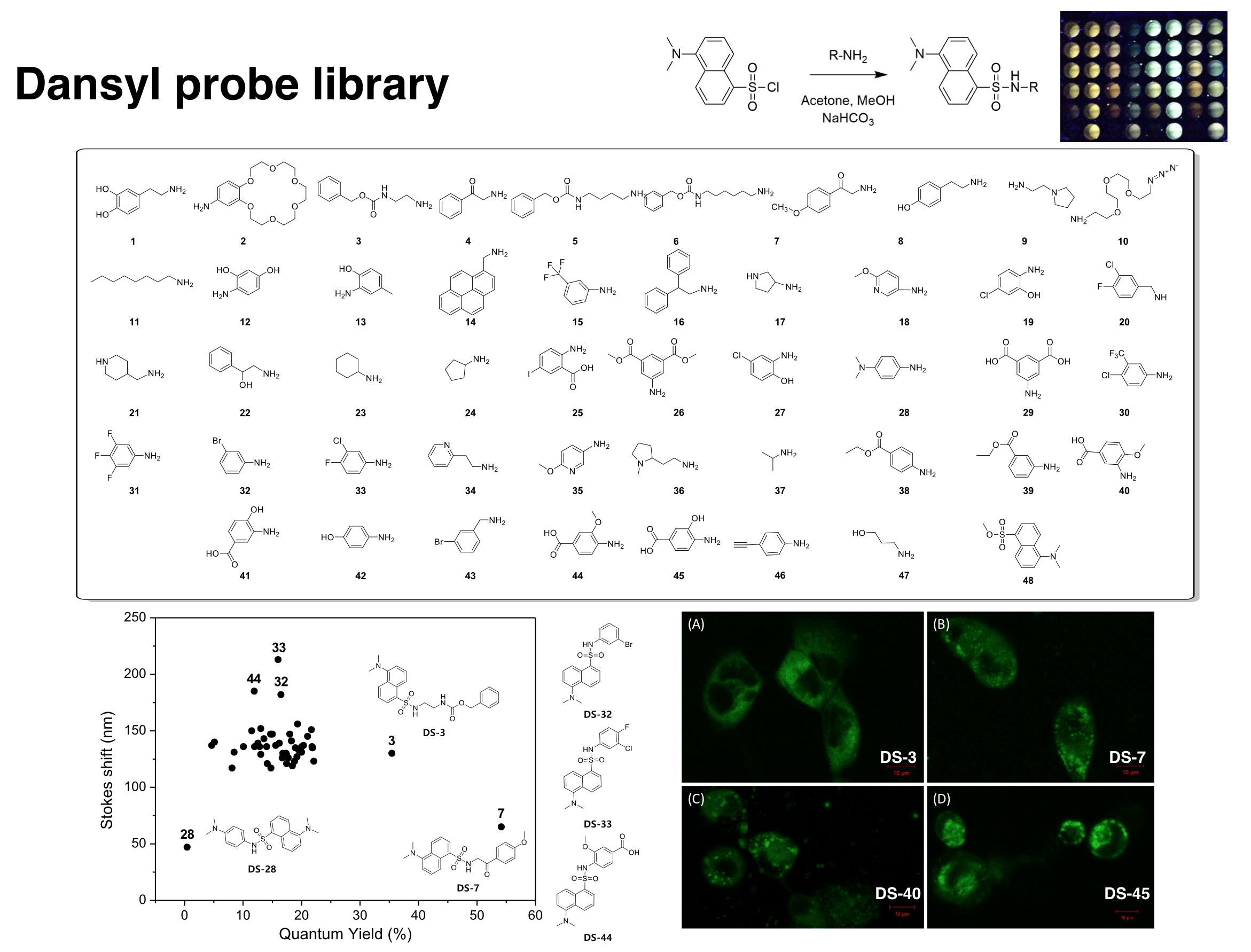
그림 3. Dansyl 구조의 형광라이브러리의 개발 및 광학특성
특히, 화학센서는 특정 분자를 인지했을 경우에만 선택적으로 색 혹은 형광세기의 변화를 신호로 발생하는 물질로, 다양한 생체분자를 표지할 형광프로브에 대한 생명과학자들의 수요는 지속적으로 증가하고 있다. 본 연구팀에서는 신규 형광프로브를 개발하기 위한 전략으로 Combinatorial Chemistry 기법을 다양한 형광 구조체에 적용하고 있다. 그 한 예로 최근 본 연구팀에서는 Dansyl 구조의 형광 라이브러리를 개발하였고, 이를 활용한 생체분자 인지 센서개발에 박차를 가하고 있다.
[연구책임자]

이
준석 교수
주소: 서울 성북구 화랑로14길 5, L2113호
한국과학기술연구원 분자인식연구센터
UST-KIST Campus 생물화학과
전화: 02-958-5092
팩스: 02-958-5059
E-mail: junseoklee@kist.re.kr
Homepage: http://leegroup.chembiol.re.kr
[연구진구성]
교수: 이준석
박사후 연구원: Dhiraj. P. Murale, Md Mamunul Haque


박사과정: 홍성철, 장세영



[대표논문]
1. Li, Z., Qian, L., Li, L., Bernhammer, J. C., Huynh, H. V., Lee, J. S., Yao, S. Q. Angew. (2016) Tetrazole
photoclick chemistry revisited: Its suitability as a bioorthogonal reaction and potential applications. Chem. Int. Ed. Engl. 55: 2002-2006.
2. Hong, S. C., Murale, D. P., Lee, J. S. (2016) Combinatorial dansyl library and its applications to pH-responsive
probes. Combi. Chem. High T. Scr. 19(5): 347-352.
3. Murale, D. P., Hong, S. C., Yun, J., Yoon, C. N., Lee, J. S. (2015) Rational design of a photo-crosslinking
BODIPY for in situ protein labeling. Chem. Comm. 51: 6643-6646.
4. Yoo, Y. H., Yun, J., Yoon, C. N., Lee, J. S. (2015) Chemical proteomic identification of T-plastin as novel host
cell response factor in HCV infection. Sci. Rep. 5: 9773.
5. Na, H., Yoo, Y. H., Yoon, C. N., Lee, J. S. (2015) Unbiased proteomic profiling strategy for discovery of b
acterial effector proteins reveals that Salmonella protein PheA is a host cell cycle regulator. Chem. Biol. 22: 453-459.
6. Lee, J. S., Lee, J. W., Kang, N., Ha, H. H., Chang, Y. T. (2015) Diversity oriented approach for
chemical biology. Chem. Rec. 15: 495-510.
7. Lee, J. S., Yoo, Y. H., Kang, J. H., Han, W. S., Lee, J. K., Yoon, C. N. (2014) Proteome reactivity profiling
for the discrimination of pathogenic bacteria. Chem. Comm. 50(33): 4347-4350.
8. Lee, J. S., Yoo, Y. H., Yoon, C. N. (2014) Small-molecule probes elucidate global enzyme activity in a proteomic
context. BMB Rep. 47(3): 149-157.