
생화학분자생물학회입니다.
유전단백체학(Proteogenomics)
작성자
관리자작성일자
2018-04-01조회수
1007유전단백체학(Proteogenomics)

박대찬
아주대학교 자연과학대학 생명과학과
dpark@ajou.ac.kr
1. 서론
빅데이터 시대가 도래함에 따라 생물학에서도 대용량 데이터 생산과 분석이 각광을 받고 있다. 분자생물학 분야에서 빅데이터를 생산하는 연구 분야를 뽑으라고 하면 각종 Omics(오믹스)가 대표적이라고 할 수 있다. Metabolomics, Lipidomics, Glycomics 등 여러 오믹스 분야가 등장하였지만, 이 중에서도 Central dogma의 핵심을 이루는 DNA, RNA, Protein 분석을 대표하는 학문이 Genomics(유전체학)와 Proteomics(단백체학)이다. 그리고 이 두 오믹스가 결합하여 Proteogenomics(유전단백체학)라는 새로운 융합 학문이 발생하였다.
유전단백체학은 Next Generation Sequencing(NGS)과 Mass Spectrometry(질량분석) 기술로부터 얻어진 유전체 및 단백체 데이터의 통합 분석을 기반으로 한다. 이 두 기술은 이미 Genome Wide, Proteome Wide한 관찰이 필요할 때 흔히 실험실에서 사용되는 도구이다. 하지만 한 연구를 위해 NGS, Mass spectrometry를 따로 둘다 적용하였다고 해서 유전단백체 연구를 하였다고 말하지는 않는다. 즉, 유전자와 단백질의 발현양을 비교하는 것은 단순한 수준에서 유전체학과 단백체학을 융합한 것이다. 유전단백체학의 정의를 정확히 이해하기 위해서는 NGS와 질량분석의 기술의 특징을 알 필요가 있다.
단백체학에서 질량분석법은 말 그대로 분자들의 질량을 바탕으로 아미노산 서열을 밝히게 된다. 질량 분석기로부터 얻어진 정보를 바탕으로 아미노산 서열을 찾기 위해서는 Spectrum을 Search할 데이터베이스가 필수적이다. 데이터베이스 없이 아미노산 서열 밝히는 High Throughput de novo Sequencing을 만들기 위해 많은 연구그룹들이 노력하고 있지만 아직은 미성숙된 기술이다. 따라서 일반적으로 대부분의 질량 분석은 표준화된 데이터베이스를 이용하게 된다. 사람의 경우에는 이미 Human Genome Sequencing이 완료되었기 때문에 어떤 아미노산 서열이 만들어질 수 있는지 추론 가능하며, 광범위하게 사용될 수 있는 Human 데이터베이스가 이용되는 것이다.
그렇다면 만약 내가 연구하고 있는 단백체의 아미노산이 표준 데이터베이스에서 존재하지 않는다면 어떻게 될까? 그 아미노산은 못 찾게 된다. 데이터 속에 있는 정보를 그냥 잃어버리게 되는 것이다. 이러한 문제를 해결하기 위해서는 샘플마다 맞춤형 데이터베이스를 만드는 것이 필요하다. 불과 10년 전만 해도 사람 개인의 유전체와 전사체 정보를 얻는 것이 비용과 시간적인 측면에서 매우 힘든 일이었다면, 지금은 NGS 기술의 등장으로 쉽게 가능해졌다. NGS는 Heterogeneous한 Nucleic Acids를 동시에 병렬적으로 Single Nucleotide 수준에서 시퀀싱하는 기술로, 맞춤형 데이터 베이스를 만들어 줄 수 있게 되었다. 따라서 유전단백체 분야는 NGS 데이터를 바탕으로 만든 유전체 데이터베이스에 질량분석 기술로 얻어진 단백체 Spectrum을 Search해서 연구하는 학문 분야이다.
생화학적 기능을 가진 분자는 대부분 단백질이기 때문에 단백체 연구에 대한 요구는 계속 증가해왔다. 하지만 증폭이 되지 않는 단백질의 특징으로 인해 그 연구의 민감도는 한계가 되어 왔고, Single Amino Acid 수준에서 대용량 시퀀싱은 개발에 여려움을 겪고 있다. 유전단백체학은 이러한 문제들을 해결할 수 있는 융복합 학문일 뿐만 아니라 개인 맞춤형 단백체 연구를 가능하게 해줄 것이다. 필자는 이 글을 통해 유전단백체의 원리와 적용되고 있는 사례에 대해 소개하고자 한다.
2. 본론
2-1. 유전단백체 분석법
유전체학 분야의 대표적인 두 기술로 Microarray와 NGS를 들 수 있다. 이 두 방식 모두 Gene Expression, Single Nucleotide Polymorphism(SNP), Copy Number Variation(CNV)를 볼 수 있다는 측면에서 유사하며 NGS만이 유일한 유전단백체 연구 도구는 아니다. 하지만 Microarray는 미리 디자인된 Probe에 대한 변이만 볼 수 있는 반면에 NGS는 유전체 또는 전사체 전체의 시퀀스를 볼 수 있다는 우월성 때문에 유전단백체 연구에 더 일반적으로 이용된다. NGS로 유전적 변이들을 대량으로 찾았을 때 이 변이들이 어떤 단백질이 될지 유추하는 것은 어렵지 않다. 아래 그림에서 보는 것처럼 NGS에서 찾아진 Mutation을 기존의 데이터베이스에 반영하여 업데이트한 후 그 시퀀스를 번역하면 되는 것이다.
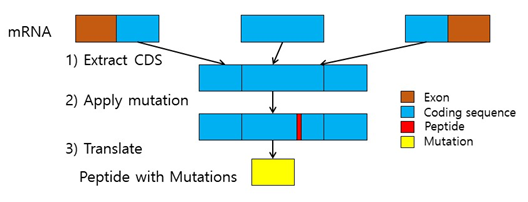
그림 1. 유전체 변이를 단백질 변이로 변형시키는 과정
이렇게 얻어진 Peptide가 실제 세포에 존재하면서 발현되고 있을지는 NGS 데이터만으로는 알 수 없다. 따라서, 질량 분석 데이터가 필요한 것이다. 질량분석 데이터는 동일한 시료에서 준비되어야만 한다. 만약 암 조직에서 DNA, RNA를 뽑아 Whole Exome Sequencing, RNA Sequencing을 했다고 가정했을 때 동일한 시료에서 단백질을 뽑아서 질량 분석을 수행할 필요가 있다. 이렇게 얻어진 유전체, 단백체 데이터는 각 분야의 정립된 분석법을 따르고 최종적으로는 유전체 데이터에서 만들어진 데이터베이스에 단백체 Spectra를 Search하게 된다. 즉, 이는 개인 맞춤형 데이터베이스를 이용하기 때문에 표준 데이터베이스에서는 찾아지지 않는 단백질 변이, 단백질 Isoform, Fusion Protein 등을 찾을 수 있게 된다.
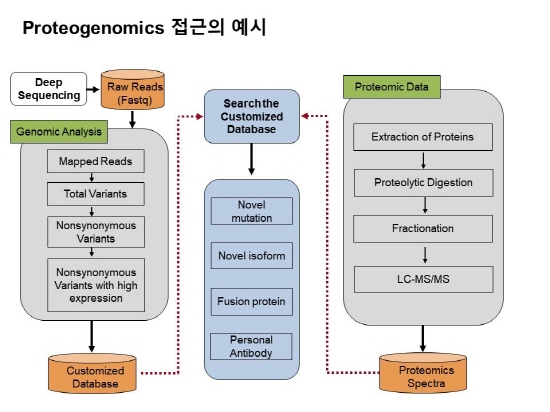
그림 2. 유전단백체 데이터 분석 흐름도
위에서 본 예는 기존의 데이터베이스에 있는 시퀀스를 업데이트하는 방식으로 유전단백체 연구가 적용된 것이다. 그렇다면 Annotation 되지 않은 새로운 단백질도 찾을 수 있을까? 맞춤형 유전단백체 분석은 새로운 ORF를 찾을 수도 있다. NGS의 RNA-seq은 de novo Sequencing을 하는 것이기 때문에 non-coding RNA(ncRNA)가 많이 발견되었으며, 다양한 종류의 5’ Untranslated Region(UTR)을 가진 mRNA도 찾아졌다. 이렇게 RNA-seq에서 찾은 Novel Transcript로 데이터베이스를 만들면 Unannotated Peptide를 찾을 수 있게 된다. 특히 Translation되었을 RNA를 찾는 실험 기술인 Ribo-seq(Ribosome에 Binding한 RNA를 Sequencing) 데이터는 Ribosome이 ncRNA와 5’ UTR에도 많이 붙는 것을 보여주었으며 그 서열을 바탕으로 Novel Peptide 후보를 예측하였다 (1). 즉, 유전단백체 접근으로 ncRNA와 5’ UTR의 용어에서 쓰인 “Non-Coding”, “Untranslated”라는 표현이 더 이상 유효하지 않은 경우를 많이 찾게 된 셈이다.
환자 특이적인 SNP를 데이터베이스에 반영하여 질량분석 Spectra를 Search하는 것은 반드시 개별 샘플 맞춤형 NGS 데이터 생산이 필요하다. 하지만 앞서 말한 ncRNA, 5’UTR에서 생성되는 Peptide를 찾기 위한 데이터베이스는 실험 없이 이론적으로 만들 수 있다. Coding, Non-Coding에 대한 Annotation을 무시하고 Human Genome 전체를 6 Frame Translation 시켜서 데이터베이스를 만든다면, 단백질이 만들어질 수 있는 모든 경우의 수로 데이터베이스를 만들 수 있게 된다. 하지만 이러한 접근은 Search Space를 지나치게 넓혀서 Spectrum Search 중 False Positive가 급격하게 증가하는 현상을 보이기 때문에, 이론적으로는 가능하지만 실제 사용하기에는 어려움이 많다. 따라서 Non-Coding Region의 Coding 가능성을 찾기 위해서는 여전히 실험적인 근거가 필요하다.
기존에 알려지지 않은 ORF를 찾기 위한 유전체 접근법으로는 앞서 소개한 Ribo-seq이 대표적이다. Ribo-seq는 새로운 Protein Coding Region을 찾게 해줄 뿐만 아니라, 기존의 Annotation된 유전자에서 Non-AUG Initiation Codon에 대한 정보도 제공한다 (1). 이외에도 새로운 UTR과 mRNA의 5’ End를 찾을 수 있는 유전체 기술로 5’ End에 Sequencing Adapter를 붙여서 RNA-seq을 수행하는 기술이 있으며 이 데이터는 새로운 Transcription Start Site를 찾는데 이용된다 (2). 한편, 단백체 접근으로도 N-Terminus 표지할 수 있으며 N-Terminus만 Enrichment시키고 나머지 단백질 부분은 제거한 이후에 질량 분석을 수행할 수 있다. 이렇게 RNA-seq으로 찾은 Novel Coding Region을 추가시킨 Search 데이터베이스를 만들고 N-Terminus Enrichment시킨 질량 분석 데이터를 Search하면 새로운 단백질 시작 부위를 찾을 수 있다 (3). 새롭게 찾아진 Micropeptide, Novel N-Terminus는 각종 질병, 세포 타입과 기능에 따라 역동적으로 조절되고 생물학적 기능이 있는 것으로 지속적으로 보고되고 있어서 앞으로 유전단백체 접근이 더욱 각광을 받을 것으로 예상된다.

그림 3. 유전단백체 연구로 Unannotated Protein을 동정
2-2. 유전단백체 연구의 작용 사례1: 암 유전체 연구
암 유전체 데이터는 대표적인 생물학적 빅데이터이다. 미국 National Cancer Institute(NCI)에서 지원하는 The Cancer Genome Atlas(TCGA) 프로젝트는 30개 이상의 암종에 대해 약 11,000여 명으로부터 나온 유전체 데이터를 약 2.5 Petabyte 축적하고 있다. 이렇게 많은 유전체 데이터를 바탕으로 수백만 개의 유전적 변이들이 보고되고 있지만 이러한 변이들이 단백질로 만들어지는지에 대해서는 아직 잘 알려지지 않았다. 이에 NCI는 2011년부터 The Clinical Proteomic Tumor Analysis Consortium (CPTAC)이라 불리는 컨소시움을 만들고 대규모 암 유전단백체 분석을 진행하고 있다. Colorectal, Breast, Ovarian에 대해서 먼저 연구를 시작하였고 2016년에는 The Applied Proteogenomics OrganizationaL Learning and Outcomes(APOLLO)와 International Cancer Proteogenome Consortium(ICPC)라는 두 개의 프로젝트를 시작하여 국제공동 연구 및 네트워크를 활발히 진행하며 데이터를 공개 및 공유하고 있다.
CPTAC은 이러한 대규모 연구를 통해 최근 100개가 넘는 Breast Cancer와 High-Grade Serous Carcinomas(HGSC) 샘플에 대해 분석하여 발표하였다 (4, 5). 흥미롭게도 유전체 데이터를 바탕으로 HGSC의 아형(Subtype)을 구분하였을 때는 4개의 아형이 관찰되었지만 단백체 데이터 기준으로는 5종의 아형이 밝혀졌다. 유방암 데이터에서도 단백체 데이터를 포함시켰을 때 새로운 아형이 찾아져서 유전체 중심의 암 아형 구분을 벗어나 새로운 아형 구분 가능성을 보여주었다. 또한, 유전체 분석이 제공해 줄 수 없는 단백체 분석의 장점에는 Posttranslational Modification과 Phosphoproteome에 대한 정보를 얻을 수 있다는 것이다. 특히 Phosphoproteome은 암세포 내 Signaling Pathway Network를 설명할 수 있는 결정적 정보로써 Gene Expression으로 주로 구성되던 Network를 더욱 정확하게 구성할 수 있게 되었다. 따라서 앞으로는 유전체 데이터 하나만으로 암의 특징을 설명하기 보다 유전단백체 통합 접근으로 더욱 정확하게 암의 특징을 이해할 수 있을 것으로 보인다.
2-3. 유전단백체 연구의 작용 사례2: 면역 레퍼토리
우리 몸에 있는 적응 면역은 T 세포와 B 세포로 이루어져 있다. 척추동물에 존재하는 이 면역 시스템은 외부 병원균에 반응할 뿐만 아니라 몸에서 생긴 질병에 대해서도 우리 몸을 보호해준다. 적응면역은 병원균 특이적인 항체(Antibody) 또는 T Cell Receptor를 생산하여 우리 몸을 보호한다. 사람이 살면서 노출되는 수많은 병원균으로부터 우리 몸을 지키기 위해서는 다양한 항체와 TCR을 만들어내는 것이 필수적이다. 이처럼 다양한 항체와 TCR의 총체를 면역 레퍼토리(Immune Repertoire)라고 부르며 이론적 다양성은 1010이 넘는다.
항체는 다양성을 확보하기 위해 항체 유전자 중 Variable, Diversity Joining 유전자들이 각 한 조각씩 DNA 수준에서 이어 붙여지게 되며, 이를 VDJ Recombination이라고 부른다. 그 이후에 Activation-Induced Cytidine Deaminase(AID)에 의해 무작위적인 방식으로 Somatic Mutation을 일으켜 그 다양성을 증가시킨다. 이렇게 만들어진 항체의 Variable Region이 Constant Region인 Immunoglobulin G, M, A, D, E 유전자를 만나 다양한 항체가 완성된다. 이 기작이 각 세포 하나에서 각각 일어나기 때문에 각 세포들은 특이적인 항체를 분비하게 되는 것이다. 이러한 세포 중 항원을 만나서 활성화되는 세포는 더 빠르게 증식하고 더 많은 항체를 만들 수 있게 된다.
TCR은 Cellular Immunity로써 세포가 직접 작동하여 기능을 하지만 B cell에서 분비된 항체는 단백질이 작동하는 Humoral Immunity다. 즉, 항체는 무리 피 속을 떠돌아다니는 단백질이기 때문에 TCR보다는 상대적으로 쉽게 우리 몸에서 항체를 추출해낼 수 있다. 그럼에도 불구하고 항체 단백질의 아미노산 서열을 읽는 것은 어려운 문제이다. 특히, 항체는 천문학적인 다양성을 지닌 단백질이기 때문에 표준 Search 데이터베이스가 존재할 수 없다. 이러한 다양한 DNA 시퀀스에 대한 맞춤형 데이터베이스를 만들기 위해서는 NGS가 가장 적합한 기술이다.
미국 University of Texas at Austin의 Georgiou 실험실은 유전단백체적 접근법을 이용하여 바이러스에 반응하는 항체 레퍼토리를 보고하였다 (6). 이 연구를 위해 건강한 사람이 독감 예방 주사를 맞기 전후에 피를 기여하였고 피에서 Peripheral Blood Mononuclear Cell(PBMC)를 분리하였다. 이 세포군에 B cell이 포함되어 있기 때문에 PBMC에서 추출한 mRNA에서 cDNA를 만든 후 항체 시퀀스를 PCR로 증폭시켰다. 이렇게 증폭된 PCR Product를 NGS 기술로 시퀀싱하여 항체 데이터베이스를 구축하였다. 단백체 데이터 생산을 위해서 동일한 사람의 피에서 항체 단백질을 Affinity Chromatography를 통해 분리하였다. 그렇게 분리된 항체 중 독감백신에 잘 붙는 항체만 다시 분리하고 Pepsin으로 Digestion을 하였다. 그 이후는 일반적인 단백체 샘플 준비와 동일하게 Trypsin으로 자른 후 질량 분석을 수행하였다. 이 데이터에서 나온 Spectra를 앞서 만들어 둔 항체 데이터베이스에 Search하여 항원 자극 전후에 어떤 변화가 있는지를 볼 수 있게 된다. 단, 이러한 접근법으로 Full-Length 항체 단백질 시퀀스를 동정할 수 있는 것은 아니며, 가장 다양성이 큰 부위인 CDR3부위의 아미노산 서열을 알고 이를 바탕으로 Clone을 정의하였다.
항체의 단백질 시퀀스 수준에서 Clone을 정의할 수 있게 됨에 따라 다양한 관찰을 할 수 있었다 (7). 첫째, 백신을 맞은 후 생성되는 항체의 60%는 백신 맞기 전에 이미 가지고 있던 항체였다. 둘째, 백신 맞기 전에 독감 항원에 대한 항체가 작을수록 백신을 맞은 후 다양한 항체가 만들어졌다. 셋째, H1 아형과 H3 아형에 각각 따로 유전단백체 연구를 하였을 때, 같은 Clone이 많이 관찰되었다. 넷째, 이처럼 Cross-Reactive 항체는 독감 바이러스를 중화(Neutralization)시키는 능력은 없으나 바이러스로부터 보호하는 기능을 있는 것으로 나타났다. 이와 같이 항체 단백질 시퀀스를 밝힐 수 있을 때 우리 몸이 외부 자극에 항체 레퍼토리가 어떻게 변화하는지 이해할 수 있었다. 더 나아가 유전단백체 접근으로 찾은 아미노산 서열로 재조합 항체를 만든다면 여러 종류의 바이러스에 대응할 수 있는 치료용 항체를 만들 수도 있을 것이다.
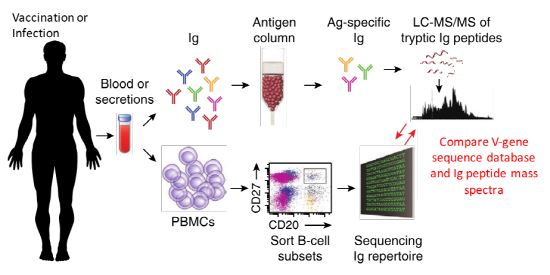
그림 4. 유전단백체을 이용한 항체 레퍼토리 연구
3. 결론
융합, 빅데이터, 맞춤형 의료는 현재 생명과학계의 큰 화두이다. 유전단백체학은 이러한 융합 접근법이 잘 반영된 연구이다. 3 Billion Base의 유전체 빅데이터와 단백체 데이터를 융합하여 맞춤형 단백질 변이와 새로운 단백질을 찾는 것이다. 대부분의 약물 타깃이 단백질인 점을 고려했을 때, 이러한 맞춤형 데이터 생산은 맞춤형 진단 또는 치료 타깃을 찾는 연구의 기반이 될 것이다. 또한 항체 레퍼토리에 적용된 유전단백체 연구는 Immunoproteogenomics라고 불릴 수 있다. 유전단백체 연구가 단순히 아미노산 변이를 찾고 발현양을 측정하는 데에만 국한되지 않고 새로운 분야에 적용된 좋은 예이다. 앞으로 유전단백체 연구가 더 많은 학문들과 융합이 되어 Proteogenomics 용어 앞에 더 다양한 분야가 추가되기를 기대해본다.
4. 참고문헌
1. Ingolia, N. T., Brar, G.A., Stern-Ginossar, N., Harris, M. S., Talhouarne, G. J., Jackson, S. E., Wills, M. R., Weissman, J. S. (2014) Ribosome profiling reveals pervasive translation outside of annotated protein-coding genes. Cell Rep 8, 1365-79.
2. Park, D., Morris, A. R., Battenhouse, A., Iyer, V. R. (2014) Simultaneous mapping of transcript ends at single-nucleotide resolution and identification of widespread promoter-associated non-coding RNA governed by TATA elements. Nucleic Acids Res 42, 3736-49.
3. Crappé, J., Ndah, E., Koch, A., Steyaert, S., Gawron, D., De Keulenaer, S., De Meester, E., De Meyer, T., Van Criekinge, W., Van Damme, P., Menschaert, G. (2015) PROTEOFORMER: deep proteome coverage through ribosome profiling and MS integration. Nucleic Acids Res 43, e29.
4. Mertins, P. et al; NCI CPTAC (2016) Proteogenomics connects somatic mutations to signalling in breast cancer. Nature 534, 55-62.
5. Zhang, H. et al; CPTAC Investigators (2016) Integrated Proteogenomic Characterization of Human High-Grade Serous Ovarian Cancer. Cell 166, 755-65.
6. Georgiou, G., Ippolito, G. C., Beausang, J., Busse, C. E., Wardemann, H., Quake, S. R. (2014) The promise and challenge of high-throughput sequencing of the antibody repertoire. Nat Biotechnol. 32, 158-68.
7. Lee, J. et al. (2016) Molecular-level analysis of the serum antibody repertoire in young adults before and after seasonal influenza vaccination. Nat Med 22, 1456-1464.
첨부파일