
생화학분자생물학회입니다.
인체 장내 마이크로바이옴 참조 유전체 데이터베이스의 개발 현황
작성자
관리자작성일자
2020-03-11조회수
1682인체 장내 마이크로바이옴 참조 유전체 데이터베이스의 개발 현황

정 해 영1,2
1메디톡스 광교R&D센터 생명정보팀
2한국생명공학연구원(KRIBB) 감염병연구센터
jeong0449@gmail.com
서론
개인의 건강이나 바이오·제약업계의 동향(특히 주가의 등락)에 조금이라도 관심이 있는 사람이라면 누구나 ‘마이크로바이옴(microbiome)’이라는 용어를 낯설지 않게 느끼는 시대가 되었다. 유산균을 먹으면 변비나 설사가 치유됨은 물론 건강에도 좋다는 전통지식의 수준을 넘어서 특정 미생물이 비만이나 대사 질환 및 아토피에 관여하고, 대변 이식을 통해 그 어떤 항생제로도 고치지 못하던 감염을 치료하며, 장내 마이크로바이옴이 면역항암제의 치료 성적을 좌우할 뿐만 아니라, 심지어 실험동물의 공포기억소거에도 관여함이 밝혀지고 있다. 이러한 연구 성과를 보면 인간의 생명현상에서 마이크로바이옴이 영향을 미치지 않는 곳이 없을 것이며, 미생물을 질환 마커나 치료제로서 이용할 수 있을 것이라는 기대를 갖는 것도 무리는 아니다. 장은 식이를 통해 섭취한 영양물질과 미생물(정착 중이었거나 또는 음식과 함께 유입)이라는 대단히 폭넓은 범위의 외부 인자에 대응하기 위하여 인체의 면역계와 신경계(enteric nervous system, ENS)가 이들과 복잡한 상호작용 벌이는 곳이기도 하다(1). 심지어 미생물과 기주 행동의 연관성을 이해하고자 미생물에 의한 신경전달물질의 생성 혹은 반응에 대한 신생 연구분야인 ‘미생물 내분비학’(microbial endocrinology)(2)이 창시되기도 하였다. 이와 같이 마이크로바이옴은 지난 십여 년간 생명과학과 의학에 종사하는 전문인력들에게는 대단한 관심과 도전 정신을 불러 일으키는 주제어가 되어 왔다.
하지만 현재 마이크로바이옴이라는 용어가 이렇게 널리 쓰이면서도 그 기원과 정확한 의미에 대해서는 아직도 논란의 여지가 있다. 어느 순간부터 마이크로바이옴의 ‘-ome’이 게놈(genome)에서 유래한 것으로 받아들여지면서, 심지어 Nature지의 마이크로바이옴 섹션1에서도 이 용어에 대하여 ‘The microbiome comprises all of the genetic material within a microbiota (the entire collection of microorganisms in a specific niche, such as the human gut).’라고 정의를 내리고 있기 때문이다. 마이크로바이오타(microbiota, 미생물총微生物叢)는 1960년대부터 이미 생태학 분야에서 쓰여온 용어였다. 흔히들 노벨상 수상자인 Joshua Lederberg가 인체의 부위에 서식하는 미생물 군집을 가리키기 위하여 2001년에 마이크로바이옴이란 용어를 처음 만들어냈다고 알려져 있지만(3) 이는 사실이 아니며(4), 당시에는 현재와 같이 유전물질이라는 개념도 여기에 녹아 들어 있지 않았다. 2001년 이전에도 이미 마이크로바이옴은 동물과 식물의 생명현상에 포함된 (미생물의) 매우 좁은 생태적 지위(ecological niche)를 의미하는 것으로 쓰여왔기 때문이다. PubMed에 등재된 문헌을 검색해 보면 이제 마이크로바이옴은 메타게놈(metagenome)을 숫적으로 능가하는 키워드가 되었으며, 메타게놈의 의미를 거의 대체하고 있다고 보아도 과언이 아닐 지경이 되었다(그림 1).
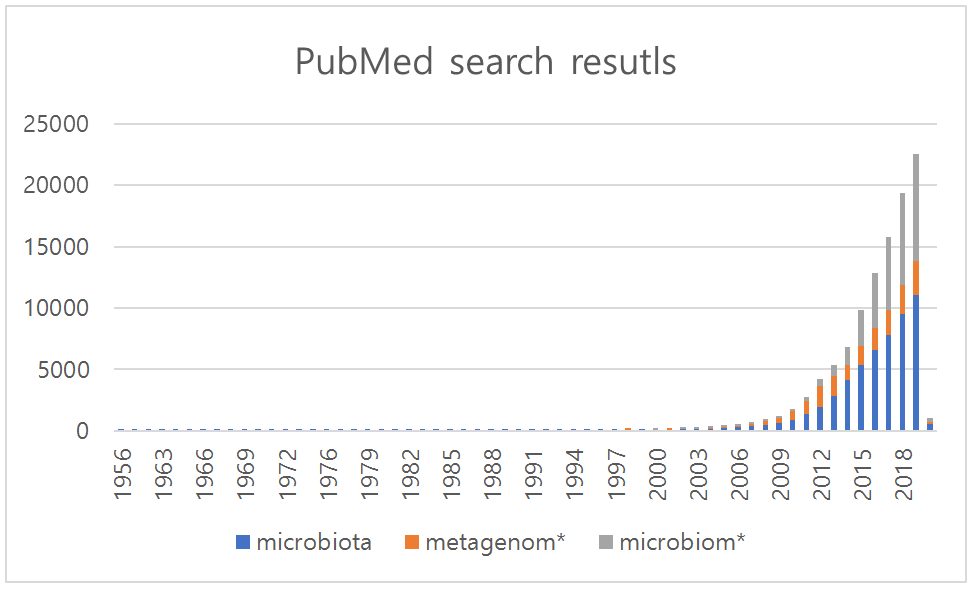
현실적으로 마이크로바이옴이라는 용어에서 집합적인 유전체의 의미를 떼어내고 원래의 쓰임새로 돌아가자고 주장할 수는 없게 되었다. 그도 그럴 것이, 21세기에 접어들면서 불어 닥친 차세대유전체염기서열해독기법(next-generation sequencing, NGS)의 혁명은 생명과학의 다른 분야와 마찬가지로 마이크로바이옴 연구의 방법론을 유전체 기반의 것으로 바꾸어 버렸기 때문이다. 즉, 순수 분리와 배양이라는 전통적인 미생물학 기법에 의존하지 않고서도 대용량 시퀀싱과 분석을 통하여 마이크로바이옴을 구성하는 미생물의 조성과 기능을 파악할 수 있게 된 것이다.
마이크로바이옴을 이해하기 위한 첫 걸음은 건강한 인체의 각 부위에 서식하고 있는 미생물의 종류를 파악하는 것으로부터 시작한다. 이는 미국 NIH가 주관한 제1기 인간 마이크로바이옴 프로젝트(Human Microbiome Project, HMP)의 주요 목표이기도 하였다. 본 글에서는 주로 장내 마이크로바이옴을 구성하는 미생물 게놈 및 유전자의 종합 카탈로그를 만들기 위한 전세계적인 노력과 그 성과에 대하여 기술하고자 한다.
본론
최신 마이크로바이옴 연구기법 – 샷건 메타게노믹스(shotgun metagenomics)
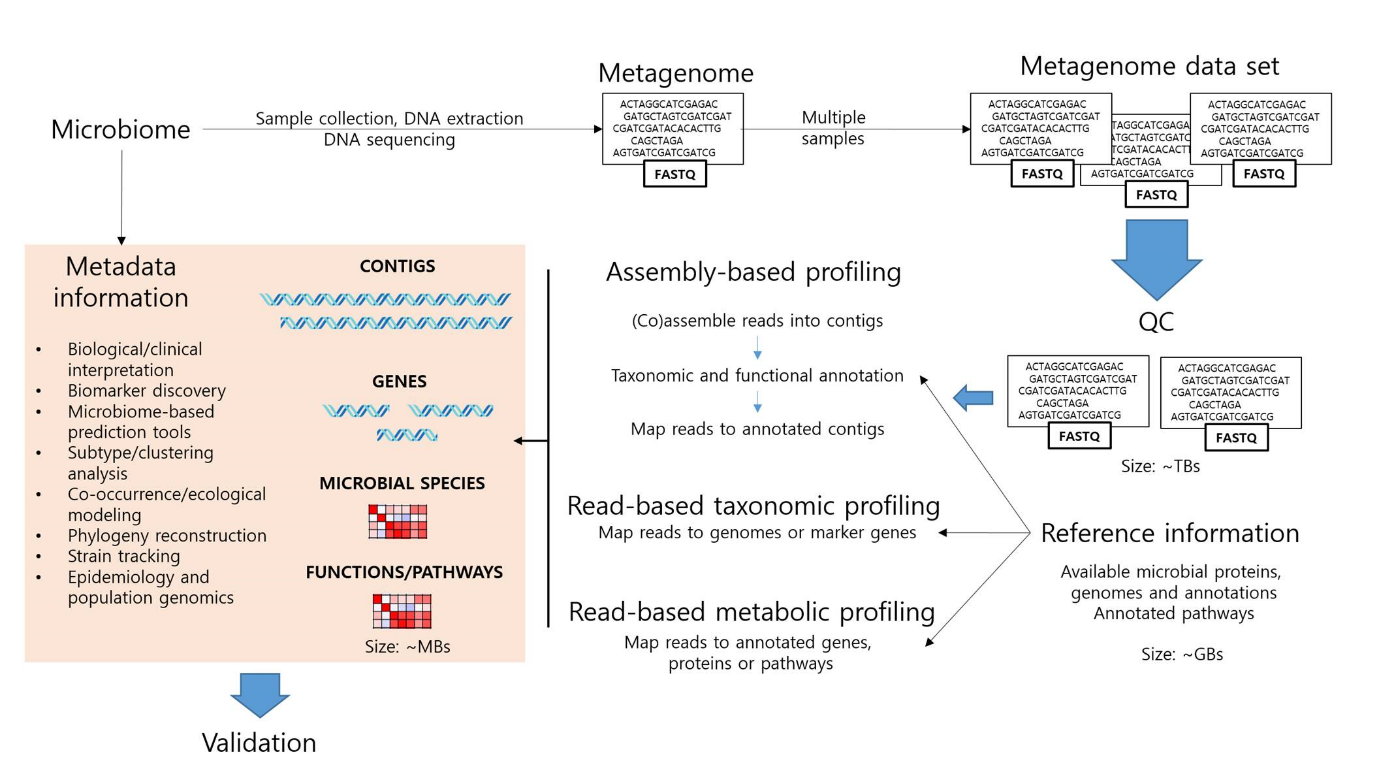
샷건 시퀀싱은 본래 단일 유전체, 즉 미생물에 한정한다면 순수 분리된 단일 균주의 유전체를 해독하기 위한 기법이었다. 배양 불가능한 미생물을 포함한 집단의 분류학적 조성을 밝히기 위하여 16S rRNA와 같은 특정 표지 유전자의 시퀀싱에 의존하던 메타게노믹스는 시퀀싱 기술과 생명정보학의 발전에 힘입어 미생물 집단으로부터 개별 균주나 심지어 단일 세포의 유전체를 재구성해내는 수준에 이르게 되었다. 이에 따라서 집단의 구성원을 구별하는 해상도는 속(genus) 수준에서 균주(strain) 단위로까지 세분화되었다. 마이크로바이옴을 대상으로 하는 연구에서 이러한 샷건 메타게노믹스는 샘플의 수집으로부터 대용량 시퀀싱 자료의 생산 및 분석에 이르는 과정을 담당하게 되었다. 좀 더 구체적으로 살펴보면, 샷건 메타게노믹스는 (i) 시료의 수집, 처리 및 시퀀싱, (ii) 시퀀싱 리드(read)의 전처리, (iii) 서열 분석을 통한 분류학적·기능적·유전체적 특성의 프로파일링, (iv) 통계적 방법을 동원한 데이터의 후처리, 그리고 (v) 확인(validation)의 다섯 단계를 거친다(그림 2)(5). 샘플과 동일한 환경에서 분리된 미생물의 참조(reference) 유전체 서열 정보는 조립 혹은 리드 기반의 분석 기법을 막론하고 분류학 및 기능 분석 과정에서 기준을 제공하게 된다. 메타게놈 데이터로부터 조립된 염기서열(metagenome assembled-genome, MAG)은 비닝(binnin)을 거쳐 개별적인 종의 수준으로 집계되는데, 배양 가능한 표준 균주가 전혀 알려지지 않은 미확인 종으로 판별되는 경우도 허다하다. MAG 조성 작업은 실제 데이터의 분석이나 인체 마이크로바이옴 기반의 참조 서열을 작성하는데 모두 중요한 과업이 된다.
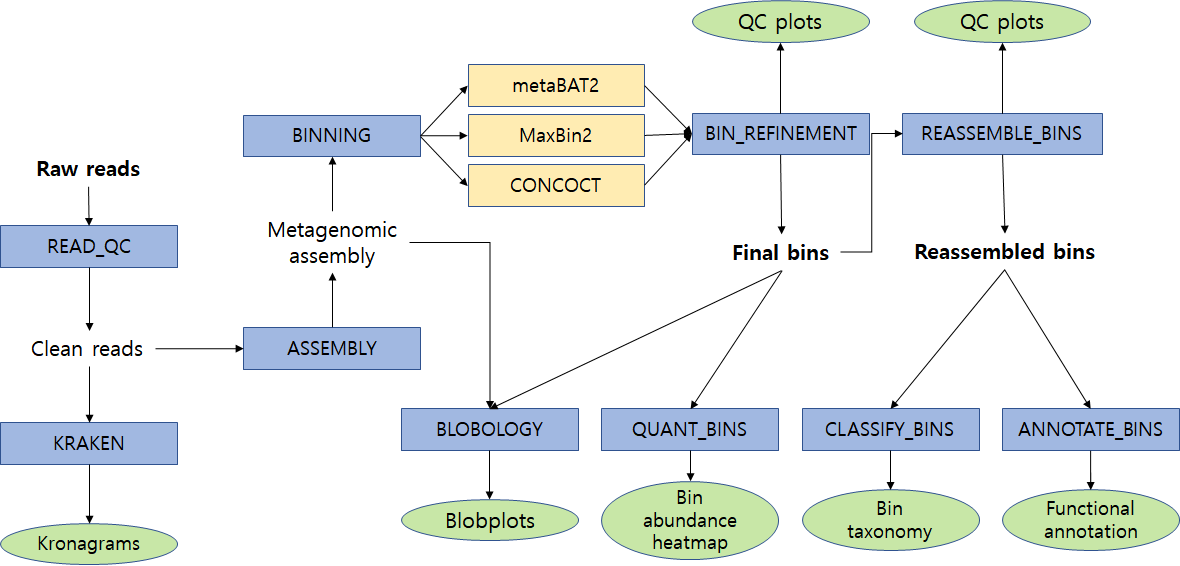
MAG 구축 작업은 데이터의 전처리, 조립, 비닝, 오염 정도 및 조립 완성도의 측정, 최종 분류 등 일련의 복잡한 과정을 거쳐 이루어지는데, 각 단계를 수행하는 개별적인 프로그램의 특성 이해와 최적화된 파라미터 결정이 관건이 된다. 이러한 용도를 위한 최신 소프트웨어 파이프라인의 사례로는 MetaWRAP(6)을 들 수 있다(그림 3).
배양 가능한 단일 균주 위주의 참조 유전체 해독
미국 국립보건원이 주도하는 인간 마이크로바이옴 프로젝트2의 1기(2007-2014) 연구 단계에서는 건강한 사람 300명의 주요 신체 부위(비강, 구강, 피부, 위장관 및 비뇨생식기)에 존재하는 마이크로바이옴을 16S rRNA sequencing과 mWGS(metagenomic whole genome shotgun) 방식으로 밝히는 것을 목표로 하였다. 이를 위해서는 같은 인체 부위 환경에서 서식하는 미생물 균주로부터 만들어진 참조 유전체 서열(reference genome sequence)에 메타게놈 서열을 매핑하는 작업이 필요하다. 인체 마이크로바이옴 점프스타트 참조균주 컨소시엄에서는 2010년 발표한 논문을 통하여 인체에서 유래한 178개 균주의 고품질 유전체 초안(draft genome)을 작성하였음을 보고하였다(7). 국제 인체 마이크로바이옴 컨소시엄(International Microbiome Consortium, IHMC)3은 1천 균주 이상의 인체 유래 미생물 유전체를 해독하는 것을 초기 목표로 설정하였으며, 이중에서 HMP 점프스타트 컨소시엄이 900건을, 유럽 연합이 지원하는 MetaHit(Metagenomics of the Human Intestinal Tract) 프로젝트가 100건을 담당하였었다. 나머지 균주에 대한 유전체 해독은 미국 에너지성에서 진행하는 Genome Encyclopedia of Bacteria and Archaea(GEBA)에서 진행되었다. GEBA의 최초 성과는 shotgun phase가 종료된 56개 균주 유전체의 초안을 2009년 발표한 것으로서(8) HMP jumpstart consortium의 논문(2010)보다 조금 더 빠르다. GEBA의 경우 초안 수준의 유전체 정보 생산을 목표로 하였지만 계통발생학적 관점에서 주의 깊게 선정된 균주에서 얻어진 것이라서 비교적 적은 수의 데이터를 투입하고서도 새롭게 발견된 단백질 패밀리의 수는 기대 이상이었음을 보고하였다. HMP 최초 논문에서는 동일 속(Lactobacillus, Bifidobacterum 및 Bacteroides)에 속하는 장내 미생물 균주를 서로 간에 1:1로 비교하여 진화적 연관성과 ANI(average nucleotide identity)를 산출하였는데, 종내(intra-specific) 균주 사이의 다양성이 기대 이상으로 높았으며 이 프로젝트를 통해 얻어진 신규 유전자가 압도적으로 높음을 알게 되었다. 즉 장내 마이크로바이옴이라는 미지의 대상을 다루게 되면서 우리가 지금까지 축적한 미생물의 다양성 지식은 아직도 얕은 수준에 머물고 있음을 의미한다. HMP 과정에서 산출된 참조 유전체 서열은 2천 건을 상회하지만(NCBI BioProject PRJNA28331), 대부분은 HMP를 위해 수집된 코호트에서 유래한 것이 아니다. NCBI에 등록된 2,309개의 유전체 서열 중 완성 또는 염색체 수준의 것은 44건에 불과하다.
중국에서는 건강한 사람의 분변에서 수집한 6천 건 이상의 세균으로부터 1,520 건의 비중복(ninredundant) 고품질 유전체 초안을 작성하고 이를 Culturable Genome Reference(CGR)라 명명하여 2019년 발표하였다(9). 그 이름에서 알 수 있듯이 배양 가능한 균주를 연구 대상으로 하였으며, 웹사이트4에서 균주에 대한 정보를 제공하고 있다. 그러나 아쉽게도 완성 유전체(assembly level: complete genome; Box 1 참조)는 아직 존재하지 않는다. 이 논문의 바로 다음 페이지에는 영국과 캐나다 및 호주의 공동 연구그룹이 구축한 Human Gastrointestinal Bacterial Culture Collection(HBC) 및 이로부터 유래한 Human Gastrointestinal Microbiota Genome Collection(HGG)을 보고한 논문이 이어지고 있다.(https://doi.org/10.1038/s41587-018-0009-7) HBC에는 237개 종(105개는 novel species)을 아우르는 737개의 균주가 수록되어 있는데, 해독된 유전체 정보는 NCBI에서 수집한 617개의 인체 위장관 관련 세균 유전체와 통합하여 HGG(1,354 유전체)를 최종적으로 구축하게 된 것이다. 이 논문에서는 HGC를 사용함으로써 샷건 메타게놈 데이터 처리에서 꼭 필요한 단계로 여겨졌던 de novo assembly 작업을 피할 수 있음을 보여주기도 하였다.
메타게놈 데이터로부터 재구성한 참조 유전체 배양 가능한 미생물을 연구에 이용한다면 균주 자원의 공유가 가능하고 실험을 통하여 미생물의 생리적 특성 및 기능에 대한 직접적인 실험적 증거를 얻을 수 있다. 그러나 현재의 기술 수준으로 순수 분리 및 배양이 불가능한 미생물이 훨씬 더 많이 존재함이 잘 알려져 있고, 배양 위주의 방법으로는 연구의 규모를 확대하는 것도 어렵다. 이를 극복하려면 샷건 메타게노믹스가 유일한 대안이며, 이를 가능케하는 생명정보학 기술은 이미 충분한 가능성을 보여주고 있다.
유럽 8개국이 공동으로 진행한 MetaHIT 프로젝트5는 점차 증가하고 있는 IBD(inflammatory bowel disease)와 비만이라는 두 주요 질환에 초점을 맞추어 장내 미생물의 유전자 및 유전체를 총망라한 목록을 작성하고, 개인별로 그것이 어떠한 차이가 있는지를 결정하며, 최종적으로는 질환 및 건강인 코호트와 연관된 유전자 및 유전체를 파악하는 것을 목표로 하였다. 2010년 발표된 첫 논문(10)에 의하면 124명의 건강인, 과체중, 비만, 그리고 IBD 환자로부터 분변 시료를 수집하여 개인당 평균 4.5 Gb의 유전체 해독을 실시하였다. 이 연구는 576.7 Gb의 염기서열을 조립하여 330만개의 비중복 유전자를 발굴하였는데, 이는 인간 유전자의 수보다 150배 정도 많은 것이다. 프로젝트에서 도출된 서열 정보의 신규성 검증에는 알려진 단일 유전체들을 참조서열로 쓰는 대신 당시 출판된 메타게놈 연구 중에서는 가장 큰 규모였던 일본인 13명 프로젝트(0.79 Gb)(11) 및 미국인 18명 프로젝트(1.83 Gb)(12)에서 생성된 리드에 매핑을 하여 비교하였다. 이로부터 밝혀진 중요한 사실 중 하나는 신규한 염기서열의 비중이 매우 높았다는 점이다.
종합 유전자 카탈로그의 등장 이상의 방법으로 만들어진 유전자 카탈로그는 발표된 이후 수년 동안 마이크로바이옴 연구의 중요한 참조 지표로 활용되었다. 서열 단편을 여기에 매핑함으로써 종 또는 유전자의 프로파일링이 가능하고, 이를 이용하여 특정 질환과 연관된 미생물의 풍부도를 찾을 수 있다(13-16). 또한 메타트랜스크립토믹스와 메타프로테오믹스가 대중화되면서 RNA 또는 단백질 데이터의 분석에도 필수적으로 쓰이게 되었다(17, 18). 하지만 참조 서열 내에 단편 혹은 키메라 형태의 유전자가 존재할 수 있고, 대부분은 단일 코호트에서 유래한 샘플을 이용하여 구성되었으며, 단백질 서열 위주의 자료라는 한계를 갖고 있다. 이를 극복하기 위하여 MetaHIT 컨소시엄과 BGI-Shenzen에서는 공개된 데이터와 새롭게 시퀀싱된 샘플을 망라하여 구축한 Integrated Gene Catalog(IGC)을 공개하였다(19). 여기에서는 기존에 공개되었던 1,018개의 샘플(중국에서 진행된 대규모 당뇨병 연구 포함)과 MetaHIT에서 새롭게 해독한 249개 샘플을 이용하여 명실상부하게 3개 대륙을 종합한 9,879,896개의 비중복 유전자 목록을 작성하였다. ICG의 작성 과정에는 NCBI와 EMBL에서 수집한 3,449개의 prokaryotic genome도 쓰였다. 정교한 선별 과정을 거쳐서 인체 장에 서식하는 것으로 판별된 983개의 유전체를 먼저 고르고, 시퀀싱 데이터의 coverage를 사용하여 511개로 줄인 다음, 이로부터 추출된 유전자를 클러스터링하여 659,492개의 유전자를 얻었다. 이를 시퀀싱 샘플의 처리를 통해 얻은 유전자와 합하여 최종 IGC 세트를 만들게 된 것이다(그림 4). IGC는 장내 마이크로바이옴의 유전자를 기준으로 한 본격적인 통합 카탈로그로는 최초의 성과물이라고 할 수 있다. IGC 자료는 별도의 웹사이트6를 통하여 공개된 상태이며, 벨기에 Ghent University의 Jeron Raes가 운영하고 있는 gut sequence database(genome sequence + gene catalog)7에서도 IGC가 중요한 비중을 차지하고 있다. |
| Box 1. Complete genomes |
| 흥미롭게도 ‘~유전체 완전 해독’이라는 과학계 기사는 흔히 접할 수 있지만 영문의 complete genome (sequence)에 해당하는 적당한 국문 용어는 잘 보이지 않는다. ‘완전 유전체’가 가장 적절할 것이나 이미 자리잡은 물리학 용어인 완전 유전체完全 誘電體(perfect dielectric)와 동음 이의어라는 문제가 있다. 필자는 개인적으로 ‘완성 유전체’라는 용어를 쓸 것을 제안한다. 여담이지만 우리나라에서는 유독 ‘전장 유전체’라는 표현도 많이 보인다. 유전체의 일부분이 아닌 전체를 다루었다는 의미의 whole-genome (sequencing)에 대응하는 것으로 파악되지만, 이는 cDNA clone 등을 수식하는 용어인 full-length를 연상하게 하므로 적절하지 않다고 생각된다. 표준 유전체라는 용어도 마찬가지이다. Reference genome은 참조의 대상이지 유전체를 정상과 비정상으로 가르는 기준이라거나 준수해야 할 표준이 아니다. 제한된 연구비를 이용하여 짧은 시간 안에 선진국 수준의 연구 성과를 도출하였다고 서둘러 발표해야 하는 국내 연구자들의 조급한 마음을 이해할 수 있으나, 과학적 기준으로 충분한 완성도를 갖추지 못한 결과물을 포장하기 위한 부정확한 용어 사용은 피하는 것이 바람직할 것이다.
|
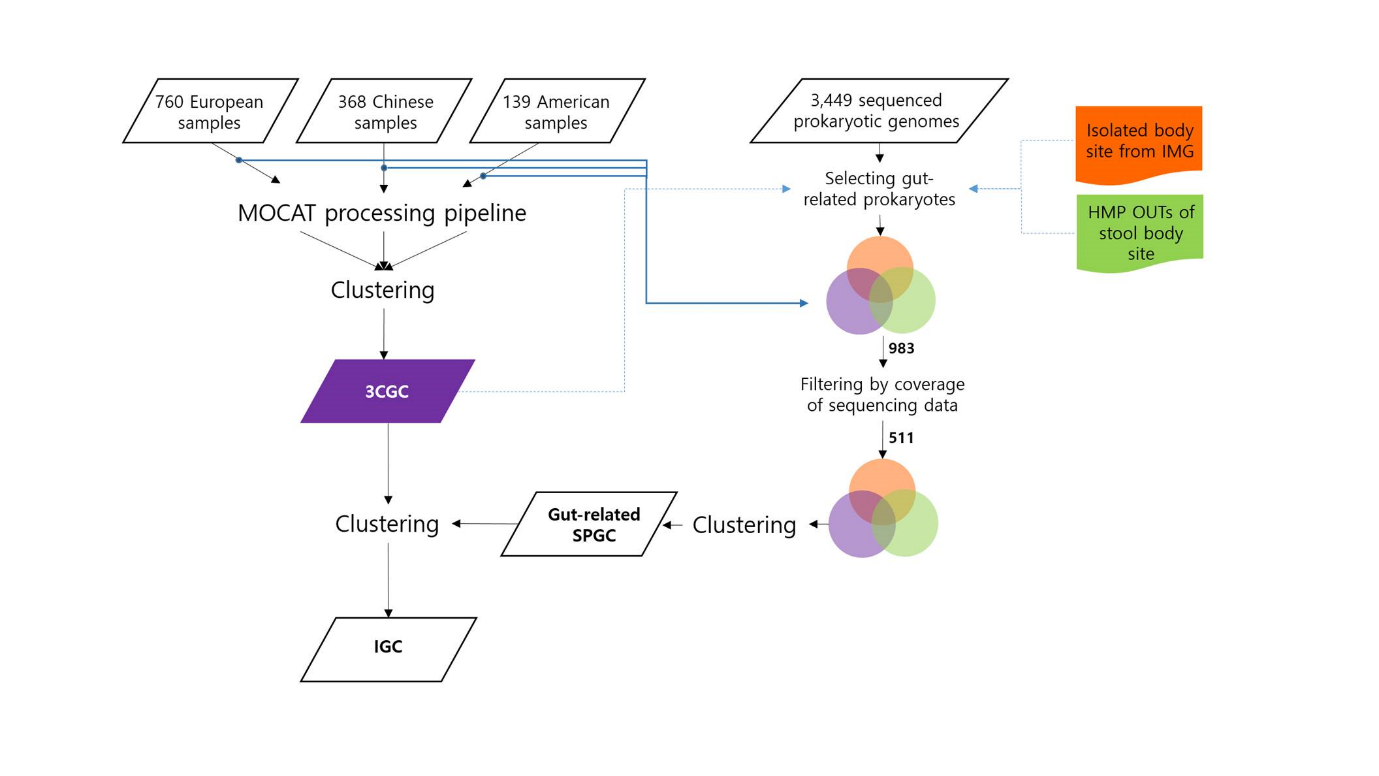
그림 4. Integrated Gene Catalog(IGC)의 구축 과정(19). MOCAT(http://mocat.embl.de/)은 메타게놈 조립과 주석화 및 프로파일링을 위한 도구이다. 클러스터링에는 CD-HIT이 쓰였다. 3CGC는 three cohorts gene catalog를, SPGC는 sequenced prokaryotic gene catalog를 의미한다.
2019년은 그동안 축적된 장내 마이크로바이옴 유래 균주 컬렉션 및 유전체 자원 연구의 대형 성과가 풍족하게 발표된 한 해였다. 앞서 언급한 두 건의 균주 컬렉션 관련 연구를 비롯하여 최소한 3 곳의 독립적인 연구 그룹이 대량의 메타게놈 시퀀싱 샘플을 이용하여 구축한 MAG 성과물을 발표하였기 때문이다. Nayfach 등 미국 연구자가 주축이 된 그룹에서는 3,810건의 분변 메타게놈 샘플로부터 60,664건의 유전체 초안을 작성하였음을 발표하였다(20). 여기에는 새롭게 확인된 2,058건의 종 단위 OUT가 포함되어 있었으며, 지금까지 알려진 시퀀싱된 장내 미생물의 다양성을 50%나 확장할 수 있었다. 영국과 호주 및 캐나다 연구 그룹에서는 11,850건의 메타게놈 샘플로부터 92,143건의 MAG를 재구성하였으며 이로부터 1,952건의 배양 불가능한 종 후보를 도출할 수 있었다(21). 모든 결과물은 웹사이트8에서 이용 가능하다.
반면 이탈리아의 Nicola Segata가 이끄는 연구 그룹에서는 앞의 것보다 한층 진일보한 인체 메타게놈 유래 유전체 정보를 발표하였다(22). 공개된 메타게놈 샘플과 새롭게 시퀀싱한 샘플(마다가스카르와 에티오피아에서 확보한 각각 112개와 50의 샘플; NCBI BioProject PRJNA485056 및 PRJNA504891)을 합친 총 9,428개의 샘플을 종합하여 154,723개의 미생물 유전체 조립물(고품질 유전체는 45%)를 재구성한 것이다. 사용된 샘플은 서로 다른 신체 부위와 연령, 국가 및 생활 스타일을 포괄하고 있다. 15만개의 유전체는 종 수준의 4,930개 species-level genome bin(SGB)를 포함하는데, 그 중에서 77%는 전혀 알려지지 않은 것이었다(unknown SGB, uSGB). uSGB는 거의 모든 샘플에 보편적으로 존재하며 특히 서구화되지 않은 인구 집단의 마이크로바이옴에서 더 큰 비중을 차지하였다. 이 연구에서 확보된 SGB과 uSGB를 사용함으로써 장 및 구강 마이크로바이옴 유래 메타게놈 서열의 매핑 가능성이 획기적으로 향상되었다. 자료는 웹사이트9에 공개되어 있다. |
| Box 2. 프리프린트(preprint) |
| 프리프린트란 심사를 거치지 않은 논문을 별도의 비용을 들이지 않고도 공개하여 인용 가능한 영구적인 DOI 번호를 부여하고 연구 커뮤니티로 하여금 정식 출판에 앞선 활용 및 피드백이 이루어지게 하는 체계인데, 다른 자연과학분야와는 달리 생명과학쪽에서는 비교적 늦게 활성화가 되었다. 많은 최고 수준 학술지에서는 프리프린트로 공개함과 동시에 학술지에 투고를 허용하고 있는만큼 이를 중복 게재나 자기 표절로 간주하지 않는다.
|
UHGG/UHGP, 인체 장내 미생물 유전체 및 유전자의 최신 컬렉션
2019년 9월 19일, 프리프린트(preprint) 서비스(Box 2)인 bioRxiv에는 바로 위에서 설명한 장내 마이크로바이옴 유래 MAG 연구자들(Philip Hugenholtz, Nicola Segata, Nikos C. Kyrpides 및 Robert D. Finn 등이 전부 주저자로 참여한 최신 논문 “A unified sequence catalogue of over 280,000 genomes obtained from the human gut microbiome”(23)이 공개되었다.
이 연구에서는 2019년 3월을 기준으로 공개된 원핵생물 유전체 중 인체 위장관에서 분리된 미생물의 것을 먼저 모은 다음 최근의 장내 미생물 컬렉션인 HBC와 CGR 유래 유전체 정보를 더하고, 마지막으로 2019년에 발표된 3건의 MAG set를 추가하여 처리함으로써 286,997건의 유전체로 구성된 Unified Human Gastrointestinal Genome (UHGG) collection을 만들어 내었다. 이로부터 추출한 단백질 서열의 집합인 Unified Human Gastrointestinal Protein(UHGP, >6억 2500만 서열) 카탈로그는 IGC에 수록된 것의 거의 두 배에 이른다. UHGG는 인류가 지금까지 만들어낸 장내 마이크로바이옴 유래 참조 유전체 컬렉션 중에는 가장 방대한 것이지만 70%의 종은 아직까지도 배양 가능한 대표 균주가 없는 실정이며, UHGP 중 40%는 신뢰할 만한 기능 주석 정보가 없다. UHGG 유전체 서열 정보는 ENA에 study accession ERP116715로 등록되었으며, UHGG, UHGP 및 SNV 카탈로그는 EBI MGnify 웹사이트10에서 이용 가능하다.
워낙 많은 종류의 유전체를 집대성한 것이기에 종내 다양성의 단면을 살펴볼 수 있다는 것이 이번 연구의 중요한 성과 중 하나이다. 분석의 정확성을 높이기 위하여 completeness가 90% 이상인 유전체 중 단일 종으로서 10개 이상의 유전체가 확보된 781개 종의 자료만을 분석한 결과 유전자들은 90% 이상의 유전체에 공통적으로 존재하는 core 혹은 rare 중 하나에 속하는 뚜렷한 bimodal distribution을 이루는 것을 관찰하였다. Core에 속하는 유전자는 핵심 대사 기능 혹은 housekeeping 기능과 깊게 연관된 반면, accessory gene은 기능이 알려지지 않았거나 mobile genetic element 및 defense mechanism(항생제 내성, CRISPR-Cas 또는 restriction-modification system)과 관련이 깊었다. SNV 분포 분석에 의하면 균주의 다양성이 대륙에 따라서 상당한 차이를 보인다는 흥미로운 결과도 도출할 수 있었다.
균주 수준 연구의 시대
방대한 메타게놈 시퀀싱 자료와 단일 유전체 자료를 통합 분석하여 얻게 된 주요한 결론은 아직도 우리가 장내 마이크로바이옴 다양성의 바다에서 최종 목적지를 찾지 못했다는 것이다. 생명정보학적 방법을 동원하여 지금까지 모르던 미생물의 유전체 서열은 입수하였지만 배양 가능한 대표 균주가 전혀 존재하지 않는 것이 대단히 많다. 또한 마이크로바이옴이 인체의 건강 상태에 미치는 영향은 많은 경우에서 종이 아니라 균주 수준에서 결정된다는 사실을 알아야 한다. 가장 가까운 사례로 대장균만 하더라도 균주에 따라서 단순한 편리공생(commensal)이나 더 바람직하게는 프로바이오틱 특성이 있는가 하면 치명적인 감염병이나 발암에 관계하는 것도 존재한다. 즉 어떤 종이 어떤 질환의 마커라는 섣부른 결론을 내리는 것은 적절하지 않으며, 균주 수준의 연관성을 찾기 위해 노력해야 한다. Segata는 2018년 mBio에 기고한 글(24)을 통해서 인체에 서식하는 미생물 집단을 ‘균주의 집합체’로 간주하는 통합적 메타게놈 연구를 진행할 것임을 선언하기도 하였다. 전통적인 배양 기법에 의존하지 않고 균주 수준의 마이크로바이옴 프로파일링 및 인체의 조건과 연관된 균주 수준의 변이체를 발굴하며 동정되지 않은 종의 균주를 재구성하는 데 필요한 기술이 충분한 수준으로 성숙하였음을 이 글에서는 밝히고 있다. 메타게놈 시퀀싱 데이터를 균주 수준에서 분석하는 소프트웨어 도구를 선택할 수 있는 폭도 과거 어느때보다 넓어졌으며(25-27), 이제는 long read 데이터를 위한 메타게놈 할당 프로그램도 개발되기에 이르렀다(28).
결론
메타게놈 기법을 동원하여 질환 혹은 건강상태를 결정하는 미생물을 찾아내고 이를 치료제 개발까지 이어 나가는 것은 현대의 마이크로바이옴 연구자라면 누구나 꾸는 꿈일 것이다. 본론에서 다룬 장내 마이크로바이옴 유래 유전자/유전체 참조 서열은 시퀀싱 데이터로부터 연구 집단 간에 유의미한 차이를 보이는 장내 미생물의 분포를 밝히는데 핵심 자료로 쓰이게 된다.
그러나 단순한 상관관계를 관찰하는 정도에 이르렀음에도 불구하고 특정 미생물이 그러한 질환 혹은 건강상태를 유발하는 인자라는 섣부른 결론을 내리는 실수를 범하기도 한다. 모든 장내 미생물이 항상 분리 및 배양 가능하지는 않다는 어려운 점이 있지만, 치료용 미생물의 개발까지 이어지기 위해서는 대상 미생물을 순수 분리 및 배양한 다음 합리적으로 설계된 실험군 및 대조군에 투여하여 이른바 개념 증명(proof of concept)에 성공해야만 한다. Patrice D. Cani는 논문 ‘Human gut microbiome: hopes, threats and promises’(29)에서 대사질환과 관련한 Prevotella copri와 Akkermansia muciniphila의 연구 사례를 통해 특정 미생물이 건강 혹은 질환 상태의 요인임을 결정하는 일이 얼마나 어려운지를 설명하였다. 균주에 따른 특성 차이 역시 상반되는 실험 결과를 설명하는데 도움을 줄 수 있을 것이며, 섭식 습관이나 약물, 장 운동성, 배변 습관 등의 환경적 요인도 마이크로바이옴의 조성에 영향을 미침을 감안해야 한다.
일회적으로 채취한 분변 샘플만으로 장내 미생물의 조성을 단순하게 판별하는 것은 더 이상 바람직하지 않다. 최근의 이미징 및 시퀀싱 기술의 발전으로 인해 미생물이 장내 공간에 대단히 복잡한 양상으로 구조화되어 분포하고 있는지를 과거 어느때보다 더 많이 알게 되었기 때문이다(30). 미생물은 장내에 결코 균일하게 존재하지 않고, 대부분은 점막층에 의해 상피세포와 물리적으로 분리되어 있으며 질환이 발생할 경우 공생 미생물은 공간적인 재배치가 일어나기도 한다.
마이크로바이옴의 유전체 정보란 대단히 중요한 기반 정보이지만 인간의 장이라는 작지만 복잡다단한 생태계를 이루는 생명개체 중 미생물을 뒤덮고 있던 미지의 베일을 한 꺼풀 벗겨낸 것에 불과하다. 이를 통하여 미생물 집단의 기본적인 속성, 즉 다양성과 대사 기능에 대한 기본적인 이해가 가능하게 되었다. 상호작용의 나머지 파트너인 인체, 그리고 양자 간에 실질적인 상호작용을 매개하는 다양한 물질 – short chain fatty acid에서 호르몬 및 세포 구성 거대 분자에 이르기까지 – 에 대한 지식도 동반하여 성장할 때 마이크로바이옴 연구의 실용화는 우리 앞에 더욱 가까운 미래로 다가올 것이다.
참고 문헌
1.
|
Yoo, B. B. and Mazmanian, S. K. (2017) The Enteric Network: Interactions between the Immune and Nervous Systems of the Gut. Immunity 46, 910-926. |
2.
|
Neuman, H., Debelius, J. W., Knight, R. and Koren, O. (2015) Microbial endocrinology: the interplay between the microbiota and the endocrine system. FEMS Microbiol Rev 39, 509-521. |
3.
|
Group, N. H. W., Peterson, J., Garges, S., Giovanni, M., McInnes, P., Wang, L., Schloss, J. A., Bonazzi, V., McEwen, J. E., Wetterstrand, K. A., Deal, C., Baker, C. C., Di Francesco, V., Howcroft, T. K., Karp, R. W., Lunsford, R. D., Wellington, C. R., Belachew, T., Wright, M., Giblin, C., David, H., Mills, M., Salomon, R., Mullins, C., Akolkar, B., Begg, L., Davis, C., Grandison, L., Humble, M., Khalsa, J., Little, A. R., Peavy, H., Pontzer, C., Portnoy, M., Sayre, M. H., Starke-Reed, P., Zakhari, S., Read, J., Watson, B. and Guyer, M. (2009) The NIH Human Microbiome Project. Genome Res 19, 2317-2323. |
4.
|
Prescott, S. L. (2017) History of medicine: Origin of the term microbiome and why it matters. Human Microbiome Journal 4, 24-25. |
5.
|
Quince, C., Walker, A. W., Simpson, J. T., Loman, N. J. and Segata, N. (2017) Shotgun metagenomics, from sampling to analysis. Nat Biotechnol 35, 833-844. |
6.
|
Uritskiy, G. V., DiRuggiero, J. and Taylor, J. (2018) MetaWRAP-a flexible pipeline for genome-resolved metagenomic data analysis. Microbiome 6, 158. |
7.
|
Human Microbiome Jumpstart Reference Strains, C., Nelson, K. E., Weinstock, G. M., Highlander, S. K., Worley, K. C., Creasy, H. H., Wortman, J. R., Rusch, D. B., Mitreva, M., Sodergren, E., Chinwalla, A. T., Feldgarden, M., Gevers, D., Haas, B. J., Madupu, R., Ward, D. V., Birren, B. W., Gibbs, R. A., Methe, B., Petrosino, J. F., Strausberg, R. L., Sutton, G. G., White, O. R., Wilson, R. K., Durkin, S., Giglio, M. G., Gujja, S., Howarth, C., Kodira, C. D., Kyrpides, N., Mehta, T., Muzny, D. M., Pearson, M., Pepin, K., Pati, A., Qin, X., Yandava, C., Zeng, Q., Zhang, L., Berlin, A. M., Chen, L., Hepburn, T. A., Johnson, J., McCorrison, J., Miller, J., Minx, P., Nusbaum, C., Russ, C., Sykes, S. M., Tomlinson, C. M., Young, S., Warren, W. C., Badger, J., Crabtree, J., Markowitz, V. M., Orvis, J., Cree, A., Ferriera, S., Fulton, L. L., Fulton, R. S., Gillis, M., Hemphill, L. D., Joshi, V., Kovar, C., Torralba, M., Wetterstrand, K. A., Abouellleil, A., Wollam, A. M., Buhay, C. J., Ding, Y., Dugan, S., FitzGerald, M. G., Holder, M., Hostetler, J., Clifton, S. W., Allen-Vercoe, E., Earl, A. M., Farmer, C. N., Liolios, K., Surette, M. G., Xu, Q., Pohl, C., Wilczek-Boney, K. and Zhu, D. (2010) A catalog of reference genomes from the human microbiome. Science 328, 994-999. |
8.
|
Wu, D., Hugenholtz, P., Mavromatis, K., Pukall, R., Dalin, E., Ivanova, N. N., Kunin, V., Goodwin, L., Wu, M., Tindall, B. J., Hooper, S. D., Pati, A., Lykidis, A., Spring, S., Anderson, I. J., D'Haeseleer, P., Zemla, A., Singer, M., Lapidus, A., Nolan, M., Copeland, A., Han, C., Chen, F., Cheng, J. F., Lucas, S., Kerfeld, C., Lang, E., Gronow, S., Chain, P., Bruce, D., Rubin, E. M., Kyrpides, N. C., Klenk, H. P. and Eisen, J. A. (2009) A phylogeny-driven genomic encyclopaedia of Bacteria and Archaea. Nature 462, 1056-1060. |
9.
|
Zou, Y., Xue, W., Luo, G., Deng, Z., Qin, P., Guo, R., Sun, H., Xia, Y., Liang, S., Dai, Y., Wan, D., Jiang, R., Su, L., Feng, Q., Jie, Z., Guo, T., Xia, Z., Liu, C., Yu, J., Lin, Y., Tang, S., Huo, G., Xu, X., Hou, Y., Liu, X., Wang, J., Yang, H., Kristiansen, K., Li, J., Jia, H. and Xiao, L. (2019) 1,520 reference genomes from cultivated human gut bacteria enable functional microbiome analyses. Nat Biotechnol 37, 179-185. |
10.
|
Qin, J., Li, R., Raes, J., Arumugam, M., Burgdorf, K. S., Manichanh, C., Nielsen, T., Pons, N., Levenez, F., Yamada, T., Mende, D. R., Li, J., Xu, J., Li, S., Li, D., Cao, J., Wang, B., Liang, H., Zheng, H., Xie, Y., Tap, J., Lepage, P., Bertalan, M., Batto, J. M., Hansen, T., Le Paslier, D., Linneberg, A., Nielsen, H. B., Pelletier, E., Renault, P., Sicheritz-Ponten, T., Turner, K., Zhu, H., Yu, C., Li, S., Jian, M., Zhou, Y., Li, Y., Zhang, X., Li, S., Qin, N., Yang, H., Wang, J., Brunak, S., Dore, J., Guarner, F., Kristiansen, K., Pedersen, O., Parkhill, J., Weissenbach, J., Meta, H. I. T. C., Bork, P., Ehrlich, S. D. and Wang, J. (2010) A human gut microbial gene catalogue established by metagenomic sequencing. Nature 464, 59-65. |
11.
|
Kurokawa, K., Itoh, T., Kuwahara, T., Oshima, K., Toh, H., Toyoda, A., Takami, H., Morita, H., Sharma, V. K., Srivastava, T. P., Taylor, T. D., Noguchi, H., Mori, H., Ogura, Y., Ehrlich, D. S., Itoh, K., Takagi, T., Sakaki, Y., Hayashi, T. and Hattori, M. (2007) Comparative metagenomics revealed commonly enriched gene sets in human gut microbiomes. DNA Res 14, 169-181. |
12.
|
Turnbaugh, P. J., Hamady, M., Yatsunenko, T., Cantarel, B. L., Duncan, A., Ley, R. E., Sogin, M. L., Jones, W. J., Roe, B. A., Affourtit, J. P., Egholm, M., Henrissat, B., Heath, A. C., Knight, R. and Gordon, J. I. (2009) A core gut microbiome in obese and lean twins. Nature 457, 480-484. |
13.
|
Qin, J., Li, Y., Cai, Z., Li, S., Zhu, J., Zhang, F., Liang, S., Zhang, W., Guan, Y., Shen, D., Peng, Y., Zhang, D., Jie, Z., Wu, W., Qin, Y., Xue, W., Li, J., Han, L., Lu, D., Wu, P., Dai, Y., Sun, X., Li, Z., Tang, A., Zhong, S., Li, X., Chen, W., Xu, R., Wang, M., Feng, Q., Gong, M., Yu, J., Zhang, Y., Zhang, M., Hansen, T., Sanchez, G., Raes, J., Falony, G., Okuda, S., Almeida, M., LeChatelier, E., Renault, P., Pons, N., Batto, J. M., Zhang, Z., Chen, H., Yang, R., Zheng, W., Li, S., Yang, H., Wang, J., Ehrlich, S. D., Nielsen, R., Pedersen, O., Kristiansen, K. and Wang, J. (2012) A metagenome-wide association study of gut microbiota in type 2 diabetes. Nature 490, 55-60. |
14.
|
Karlsson, F. H., Tremaroli, V., Nookaew, I., Bergstrom, G., Behre, C. J., Fagerberg, B., Nielsen, J. and Backhed, F. (2013) Gut metagenome in European women with normal, impaired and diabetic glucose control. Nature 498, 99-103. |
15.
|
Le Chatelier, E., Nielsen, T., Qin, J., Prifti, E., Hildebrand, F., Falony, G., Almeida, M., Arumugam, M., Batto, J. M., Kennedy, S., Leonard, P., Li, J., Burgdorf, K., Grarup, N., Jorgensen, T., Brandslund, I., Nielsen, H. B., Juncker, A. S., Bertalan, M., Levenez, F., Pons, N., Rasmussen, S., Sunagawa, S., Tap, J., Tims, S., Zoetendal, E. G., Brunak, S., Clement, K., Dore, J., Kleerebezem, M., Kristiansen, K., Renault, P., Sicheritz-Ponten, T., de Vos, W. M., Zucker, J. D., Raes, J., Hansen, T., Meta, H. I. T. c., Bork, P., Wang, J., Ehrlich, S. D. and Pedersen, O. (2013) Richness of human gut microbiome correlates with metabolic markers. Nature 500, 541-546. |
16.
|
Nielsen, H. B., Almeida, M., Juncker, A. S., Rasmussen, S., Li, J., Sunagawa, S., Plichta, D. R., Gautier, L., Pedersen, A. G., Le Chatelier, E., Pelletier, E., Bonde, I., Nielsen, T., Manichanh, C., Arumugam, M., Batto, J. M., Quintanilha Dos Santos, M. B., Blom, N., Borruel, N., Burgdorf, K. S., Boumezbeur, F., Casellas, F., Dore, J., Dworzynski, P., Guarner, F., Hansen, T., Hildebrand, F., Kaas, R. S., Kennedy, S., Kristiansen, K., Kultima, J. R., Leonard, P., Levenez, F., Lund, O., Moumen, B., Le Paslier, D., Pons, N., Pedersen, O., Prifti, E., Qin, J., Raes, J., Sorensen, S., Tap, J., Tims, S., Ussery, D. W., Yamada, T., Meta, H. I. T. C., Renault, P., Sicheritz-Ponten, T., Bork, P., Wang, J., Brunak, S., Ehrlich, S. D. and Meta, H. I. T. C. (2014) Identification and assembly of genomes and genetic elements in complex metagenomic samples without using reference genomes. Nat Biotechnol 32, 822-828. |
17.
|
Xiong, X., Frank, D. N., Robertson, C. E., Hung, S. S., Markle, J., Canty, A. J., McCoy, K. D., Macpherson, A. J., Poussier, P., Danska, J. S. and Parkinson, J. (2012) Generation and analysis of a mouse intestinal metatranscriptome through Illumina based RNA-sequencing. PLoS One 7, e36009. |
18.
|
David, L. A., Maurice, C. F., Carmody, R. N., Gootenberg, D. B., Button, J. E., Wolfe, B. E., Ling, A. V., Devlin, A. S., Varma, Y., Fischbach, M. A., Biddinger, S. B., Dutton, R. J. and Turnbaugh, P. J. (2014) Diet rapidly and reproducibly alters the human gut microbiome. Nature 505, 559-563. |
19.
|
Li, J., Jia, H., Cai, X., Zhong, H., Feng, Q., Sunagawa, S., Arumugam, M., Kultima, J. R., Prifti, E., Nielsen, T., Juncker, A. S., Manichanh, C., Chen, B., Zhang, W., Levenez, F., Wang, J., Xu, X., Xiao, L., Liang, S., Zhang, D., Zhang, Z., Chen, W., Zhao, H., Al-Aama, J. Y., Edris, S., Yang, H., Wang, J., Hansen, T., Nielsen, H. B., Brunak, S., Kristiansen, K., Guarner, F., Pedersen, O., Dore, J., Ehrlich, S. D., Meta, H. I. T. C., Bork, P., Wang, J. and Meta, H. I. T. C. (2014) An integrated catalog of reference genes in the human gut microbiome. Nat Biotechnol 32, 834-841. |
20.
|
Nayfach, S., Shi, Z. J., Seshadri, R., Pollard, K. S. and Kyrpides, N. C. (2019) New insights from uncultivated genomes of the global human gut microbiome. Nature 568, 505-510. |
21.
| Almeida, A., Mitchell, A. L., Boland, M., Forster, S. C., Gloor, G. B., Tarkowska, A., Lawley, T. D. and Finn, R. D. (2019) A new genomic blueprint of the human gut microbiota. Nature 568, 499-504. |
22.
| Pasolli, E., Asnicar, F., Manara, S., Zolfo, M., Karcher, N., Armanini, F., Beghini, F., Manghi, P., Tett, A., Ghensi, P., Collado, M. C., Rice, B. L., DuLong, C., Morgan, X. C., Golden, C. D., Quince, C., Huttenhower, C. and Segata, N. (2019) Extensive Unexplored Human Microbiome Diversity Revealed by Over 150,000 Genomes from Metagenomes Spanning Age, Geography, and Lifestyle. Cell 176, 649-662 e620. |
23.
| Almeida, A., Nayfach, S., Boland, M., Strozzi, F., Beracochea, M., Shi, Z. J., Pollard, K. S., Parks, D. H., Hugenholtz, P., Segata, N., Kyrpides, N. C. and Finn, R. D. (2019) A unified sequence catalogue of over 280,000 genomes obtained from the human gut microbiome. bioRxiv, 762682. |
24. | Segata, N. (2018) On the Road to Strain-Resolved Comparative Metagenomics. mSystems 3. |
25.
| Nayfach, S., Rodriguez-Mueller, B., Garud, N. and Pollard, K. S. (2016) An integrated metagenomics pipeline for strain profiling reveals novel patterns of bacterial transmission and biogeography. Genome Res 26, 1612-1625. |
26.
| Costea, P. I., Munch, R., Coelho, L. P., Paoli, L., Sunagawa, S. and Bork, P. (2017) metaSNV: A tool for metagenomic strain level analysis. PLoS One 12, e0182392. |
27.
| Truong, D. T., Tett, A., Pasolli, E., Huttenhower, C. and Segata, N. (2017) Microbial strain-level population structure and genetic diversity from metagenomes. Genome Res 27, 626-638. |
28.
| Dilthey, A. T., Jain, C., Koren, S. and Phillippy, A. M. (2019) Strain-level metagenomic assignment and compositional estimation for long reads with MetaMaps. Nat Commun 10, 3066. |
29. | Cani, P. D. (2018) Human gut microbiome: hopes, threats and promises. Gut 67, 1716-1725. |
30.
| Tropini, C., Earle, K. A., Huang, K. C. and Sonnenburg, J. L. (2017) The Gut Microbiome: Connecting Spatial Organization to Function. Cell Host Microbe 21, 433-442. |
첨부파일