
생화학분자생물학회입니다.
숭고한 희생정신으로 기증된 시신으로 사람 세포 Lineage tracing을 연구하는 의과학자
작성자
관리자작성일자
2019-11-11조회수
5879
숭고한 희생정신으로 기증된 시신으로
사람 세포 Lineage tracing을 연구하는 의과학자

오지원
경북대학교 의과대학 해부학교실
ohjiwon@knu.ac.kr
1. Lineage tracing 연구
한 개체 내에서 각각의 기능으로 분화한 세포의 원류를 찾는 것은 줄기세포 연구에서 아주 중요한 과업중에 하나라고 볼 수 있다. 특히, 줄기 세포의 경우에는 각각의 세포가 어떤 경로로 특정 장기의 세포로 분화가 되는지를 아는 것이 기능적인 측면에서나 재생의학적인 측면에서 아주 중요하다고 볼 수 있다. 특정 줄기 세포에서 분열되어 이 세포가 어디에 분포하고, 어떤 기능적인 역할을 하는지를 추적 연구하는 것을 통칭하여 Lineage Tracing 연구라고 한다.
Lineage tracing 연구의 시작을 알리는 “Cell-lineage”의 개념을 이해하려면, 1892년도로 거슬러 올라가야 한다. Edmund Wilson은 Annelid body(환충)를 분석하면서, 특정 세포들이 연속적으로 분열하면서 각각의 “운명(Fate)”이 있는 것 같다고 보고를 한다(1). 이 개념은 1905년도에 확장되어 Cell lineage에서 Invariant와 non-invariant 세포라는 개념을 형성하였고 이는 현미경을 이용한 Ascidian egg(해초강)의 발생시기에 대한 관찰로 증명이 되었다(2).
Lineage tracing 연구는 다양한 분자의학적 기술 개발과 함께 생물학을 이끌었다고 해도 과언이 아닐정도로 새로운 연구 기법과 생물학적 개념을 도입하였다. 특히, 2018년도에 작고한 John Sulston경은 광학적 방법으로 C.elegans (예쁜 꼬마 선충)을 관찰하여 Apoptosis라는 새로운 개념을 도입하였고, 이는 결국 노벨 생리의학상 수상으로 이어졌다.(3, 4) 또한, Cre-LoxP를 통한 각 세포가 가진 DNA를 영구적으로 변형시켜 “낙인”을 찍는 lineage tracing 기법은 학문적으로 기능적 세포 분화 연구에 획기적인 도구적 발판을 마련하였고(5), 현재까지도 생물학적 인과관계 측면에서 절대적 도구로 이용되고 있다. 최근 CRISPR-Cas9 유전자 편집을 적용하여 genetic scar를 만든 후, Massive NGS를 통해 세포의 일생을 재구성한 lineage tracing 기법은 다양한 동물 모델에서 발생 과정에 대한 이해의 폭을 확연히 넓혀 주었고, 현재 발생학, 줄기세포 연구의 큰 흐름이라고 볼 수 있다. (6-8)
Lineage tracing을 이용한 연구 방법은 확실한 과학적 증거를 제시하기 위해서 보다 엄밀한 방법을 사용하는 방향으로 발전하였는데, 최초의 연구들은 현미경으로 단순 관찰한 것으로 충분하였으나, 최근에는 세포 수준의 DNA 레벨에서의 증거 없이는 Lineage를 확실하게 증명할 수 없을 정도이다. 이런 관점에서 Cre-LoxP를 이용한 연구방법론과 CRISPR-Cas9 시스템을 이용한 방법들과 같이 유전자를 변형하거나 조작한 동물들을 이용한 연구들이 Lineage Tracing 연구의 대세가 되었다. Brainbow mouse, Confetti mouse, Developmental barcode, Zebrafish에 이용된 GESTALT 기술들이 그 예시라고 할 수 있다. (9-10)
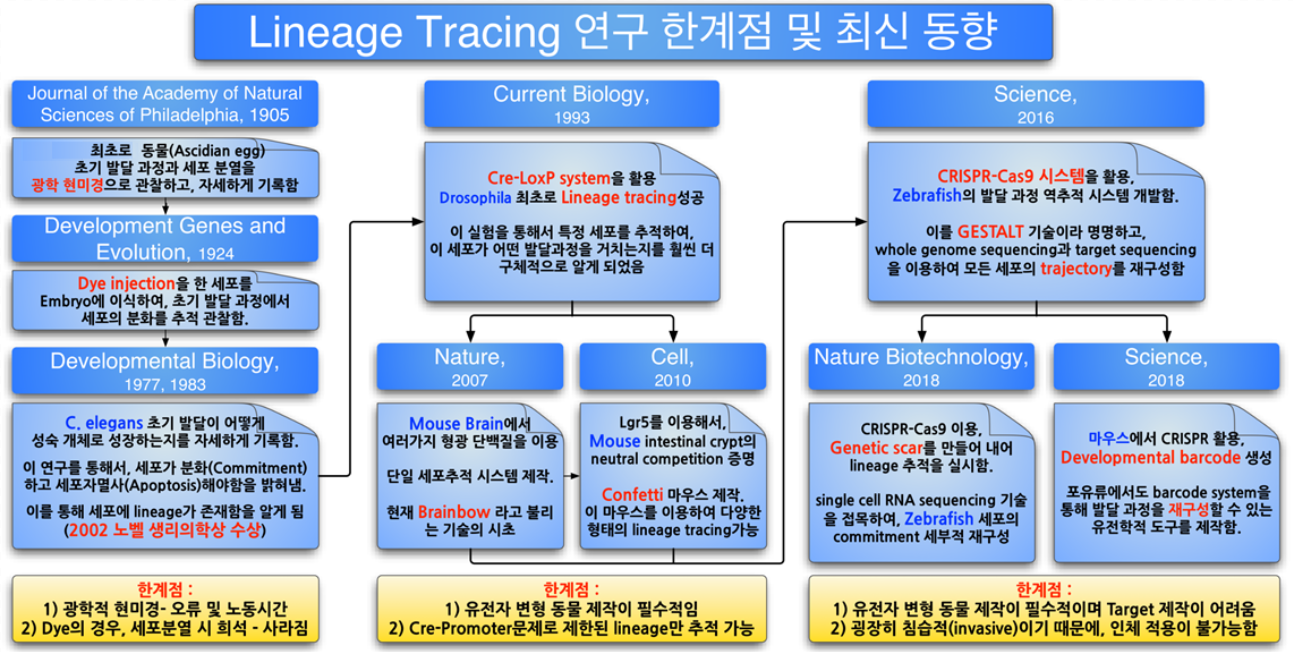
Figure 1 Lineage Tracing 연구의 한계점과 동향
하지만, 위 방법론들은 필연적으로 살아있는 세포 혹은 개체 수준에서 유전자 변형 및 조작을 해야한다는 한계가 있으며, 이는 이미 태어난 사람에게는 절대 적용할 수 없다는 것은 자명하였다. 사람의 발생과정은 동물의 발생과정과 유사하다고는 알려져 있으나, 이를 학문적 동치로 볼 수 없기 때문에, 보다 확실한 증거를 이용해서 사람의 발생과정을 포함하여, 사람 세포의 Lineage tracing을 연구하고자 하는 수요가 늘어났다. 하지만, 한동안은 기술적인 한계로 윤리적인 문제를 해결하면서 동시에 과학적 증거를 제시하는 것이 불가능하다고 여겨졌었다.
2. Next generation sequencing의 발전과 somatic variants(체성 돌연변이)를 이용한 Lineage tracing
Next generation sequencing(NGS)의 발전은, 우리가 생명체에 대해서 몰랐던 사실에 대해서 많은 것을 알게해 주었다. NGS의 발전은 다양한 의과학 분야에서의 발전을 이끌었지만, 특히 암연구에서 가장 많은 발전을 이루었다고 해도 과언이 아니라고 할 수 있다. 왜 암이 생기는지, 그리고 암이 어떤 돌연변이에 의해서 발생하는지에 대한 연구는 암 연구자들이 가진 가장 근원적인 질문이였지만, NGS의 발전으로 비로소 Whole genome 수준에서 암을 분석할 수 있게 되었다. 전통적인 연구들을 통해서 이미 알려진 tumor suppressor gene이라든지, oncogene들 뿐만 아니라, 다양한 유전자들이 주도적으로 암을 유발할 수 있을 것이라고 예측되었다. 이들 유전자의 돌연변이들을 통칭하여 Driver mutation이라고 부르게 되었는데, 이 개념을 통해서 암에서 이들이 어떻게 정상 세포에서 종양 세포로 변화되는지에 대한 연구가 가속화되었다.(11)
하지만, NGS를 통해서 다양한 암을 분석해보니, 주요 유전자에서만 돌연변이가 생기는 것이 아니라, Whole genome 전반에 걸쳐서 돌연변이가 생긴 흔적들이 존재하였다. 1989년도 PNAS에 나온 논문에서는 Astrocytoma에서 17번 염색체를 활용해, non-coding region의 다양한 부위에도 돌연변이가 존재함을 보고하였고(12), Driver 뿐만 아니라, 다양한 돌연변이들이 암세포에 퍼져있음을 확인하는 다양한 결과들이 나오게 되었다. 현재까지 이들 돌연변이들의 기능이 확실히 밝혀지지 않았으면서도, 상대적으로 이들의 수가 상대적으로 많기 때문에, Driver mutation과는 대비되는 개념의 Passenger mutation이라고 부르고 있다. 일부 그룹에서는 passenger mutation이 미치는 암 발생과 예후 등에 미치는 영향에 대해서 연구하고 있다.(13)
이렇게 다양한 돌연변이가 암세포에 존재함을 NGS를 통해 알게되면서, 사람들은, 재미난 도전을 해보기로 마음을 먹었다. 돌연변이가 딸세포에 전달된다는 단순한 사실을 이용하여, 돌연변이를 통해서 암세포의 “인생사”를 재구성해보겠다는 야심찬 도전이 시작된 것이다. 그리고 이들의 도전은 2012년 The New England Journal of Medicine(NEJM)에 보고되었다.(14) 이들은 Intratumor heterogeneity를 알아보기 위해서, 각 암세포들이 가지고 있는 체성 돌연변이(somatic variants)를 활용하여, 이들의 Phylogenetic tree를 그렸다. 그리고 체성 돌연변이의 공유된 정도와 시기에 근거하여, 정상 조직에서 암으로 진행되는 과정을 역추적하고, 이들의 전이 과정을 재구성하였다. 비로소 윤리적인 문제 없이 사람 세포를 In vivo 수준에서 Lineage tracing할 수 있는 방법론이 개발된 셈이었다. 이 논문은 2019년 11월 현재, 2012년도에 보고된 이후, 7년동안 5600회 이상 인용이 되었다. 이 논문 이후 다양한 보고들이 이와 비슷한 방법론을 적용하여 사람 종양 세포의 역사를 재구성하였고, 이들이 가지는 cancer cell heterogeneity를 활용하여 항암제 치료 감수성이 다를 수 있음을 예상하고 이를 실험적으로 증명하였다. (15-17)
그렇게 NGS를 통해 많은 암 연구가 진행되면서, 새로운 사실을 알게되었다. 일반적으로 암세포의 경우에는 Driver mutations 때문에, DNA repair 기능이 작동이 잘 안되어서 새롭게 돌연변이의 발생 빈도가 늘어난다고 알려져 있었다. 그에 반해 정상 세포의 경우에는 mosaicism과 같은 특수한 질환을 제외하고는 체성 돌연변이가 거의 없다고 생각했었는데, 의외로 정상 세포에서도 생각보다 많은 돌연변이가 있다는 보고들이 나오기 시작한 것이다. 특히, 수정체에서 시작하여 Inner cell mass까지 진행되는 Early embryonic stage에서 이들의 빈도가 생각보다 많이 일어난다는(0.1-3개) 보고가 나오기 시작하였다. (18-21)
만약 세포 분열마다 0.1-3개의 돌연변이가 축적된다면, 세포분열을 거듭할수록 한 개체가 가진 모든 세포는 분열에 따라 서로 다른 돌연변이가 축적되기 때문에, 서로 다른 DNA를 가지고 있게 된다. 결과적으로, 한 명의 인간은 대략 30조개의 세포를 가지고 있는데, 이들 30조개의 세포는 평생을 거치면서 수천 번 이상 분열하기 때문에, 분열시마다 축적된 돌연변이는 결국, 세포마다 고유의 DNA패턴을 가지는 것으로 귀결된다고 추론할 수 있다.
3. 시신에서 배양한 세포를 통한 사람 세포 Lineage tracing 연구
사람을 포함한 대부분의 동물은 발생과정에서 Ectoderm(외배엽), Mesoderm(중배엽), Endoderm(내배엽)으로 구분되어 분화되며, 이들은 고유의 발생-분화과정을 거쳐서 특정 조직이 된다. 세포의 일생을 올바르게 재구성하려면, 한 개체 내에서 이들 세 가지 배엽에서 유래한 세포를 확보해야만 이들의 과정을 재구성할 수 있다. 예컨대, 외배엽 유래의 각막 세포와 중배엽 유래의 혈액 세포, 그리고 내배엽 유래의 대장 세포를 동시에 한 개체에서 확보해야만, 각각의 배엽 유래의 세포가 한 개의 조상 세포를 가졌는지, 아니면 다양한 조상 세포가 있는지를 확인할 수 있고 엄밀한 의미에서 Early postzygotic lineage tracing을 수행할 수 있다.
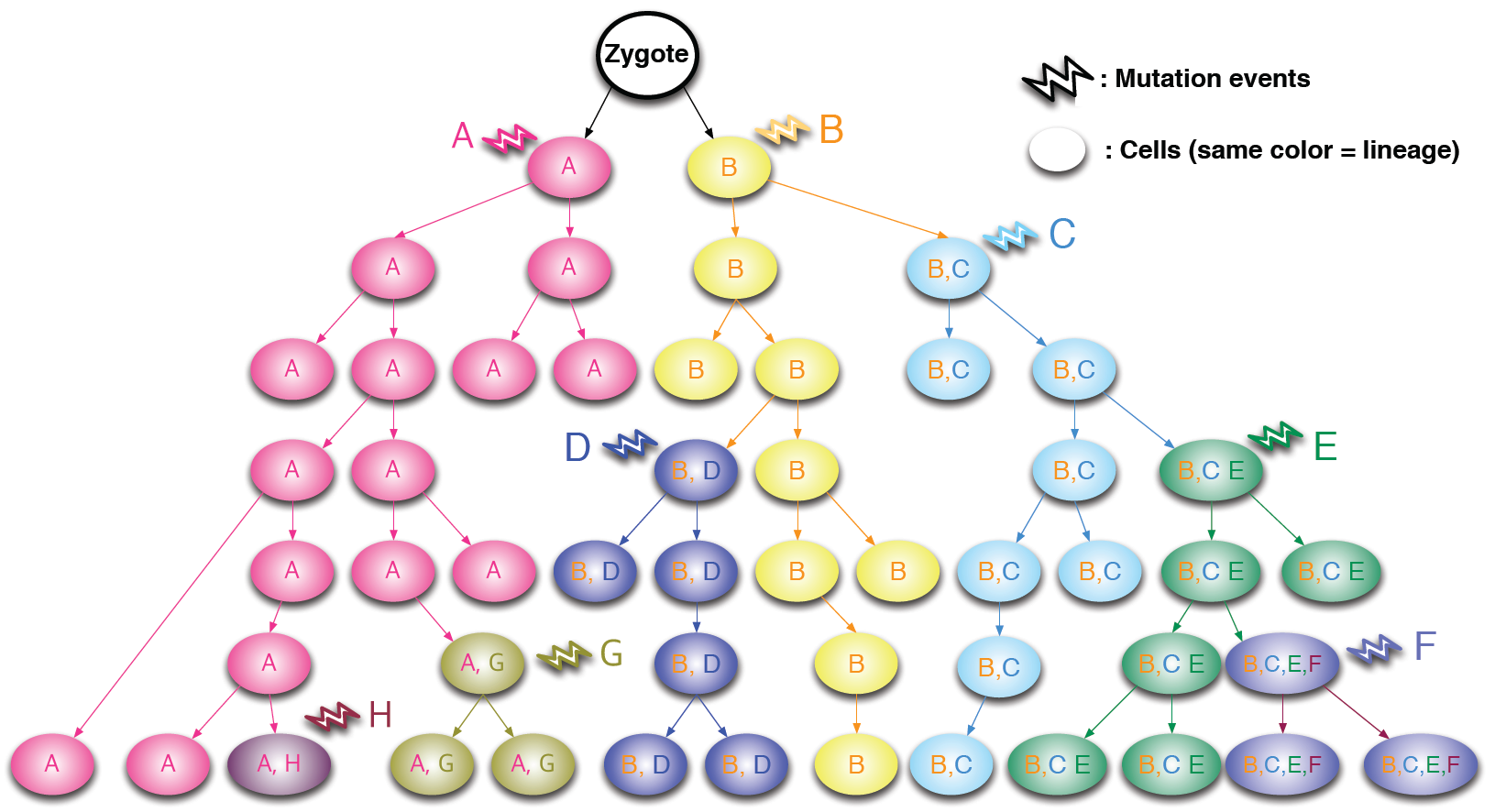
Figure 2 가상의 세포분열과 돌연변이 축적 과정
(adapted from Analysis of cells’s and nuclei’s genome by Next Generation Sequencing, in press)
일반적인 의과학 연구에서 확보되는 수술 환자 샘플들은, 위 세 가지 배엽의 세포를 동시에 확보하기가 상당히 어렵다. 그에 반해, 기증된 시신의 경우에는 인체의 육신을 온전히 가지고 있기에, 한 개체 내에서 이들 3가지 배엽 유래의 세포를 충분히 확보할 수 있으며, 이는 해부학 교실이 가진 가장 큰 강점이라 할 수 있다. 물론, 시신의 경우에는 “사람”이 아닌 것으로 간주되어, 기존 환자 동의 IRB와는 체계가 다른 형태로 진행되기에 현존하는 시체해부보존법 법령의 중간지대에 있어 행정적인 어려움이 있는 건 사실이다. 특히, 응급으로 발생하는 시신 기증에 있어서, 한 구의 시신에서 200개 이상의 서로 다른 세포를 배양하는 것은 세포 배양 프로토콜뿐만 아니라, 유가족 동의 등의 사회 문화적인 부분과도 연계가 있기 때문에, 상당히 고려할 부분들이 많이 존재하는 것 역시 사실이다.
그럼에도 불구하고, 시신을 이용하여 배양된 세포 연구는 사람 발생학 연구의 윤리적인 문제를 해결하고, 정상 세포가 가진 돌연변이의 축적 history를 확실히 알 수 있는 방법 중 하나라고 볼 수 있다. 다양한 난관이 있는 것은 사실이지만, 현재 실험실의 8명의 연구원들과 함께 극복해 나가고 있으며, 많은 수의 열정적인 공동 연구자와 함께 문제를 해결하고 있다.
해부학 교실에서 약 4년간 근무하면서, 의대생과 대부분의 의사들, 그리고 생명과학 연구자들이 해부학을 “오래된” 학문으로 평가하는 것에 개인적인 아쉬움이 있다. 최근 들어 기초의학 교실의 외연 구분이 모호해진 것은 분명하지만, 최신 실험기법들이 등장하여 연구의 트렌드를 이끄는 것처럼 보이는 생화학, 생리학, 면역학, 분자의학 등은 많은 연구자들이 “변화하는 최신 학문”으로 인식하는데 반해, “해부학”은 예전부터 있어 왔기 때문에, 그리 큰 변화가 없는 오래된 학문으로 인식하는 태도가 만연한 것 같다. 하지만, 이에 대해서 개인적으로는 동의할 수 없고, 장기적으로 해부학에 대한 인식을 변화시키고자 하는 의지가 큰 편이다.
해부학을 연구하는 학자로서 장기적으로 하나의 바람이 있다면, 현재 해부학을 바라보는 연구자들과 의사들의 시선을 획기적으로 변화시키고 싶다. 특히, 시신을 단순히 의대 교육용이나 외과 의사들의 임상해부학 수련용으로 보는 것이 아니라, 그 사람이 평생 동안 살아온 세포의 역사를 온전히 보전한 “인체 조직”이라는 패러다임 변화를 통해, 인체에 숨겨진 비밀을 밝혀내는 것이 개인적인 학문 목표이다. 조금만 더 관점을 달리 보면, 기증된 시신은 생이 끝났다는 점을 제외하고는 수술을 통해 얻는 조직과는 수준이 다른, 모든 배엽의 세포들을 온전히 구할 수 있는 사람 조직과 세포의 모음인 셈이고, 숭고한 기증 정신에서 나온 자기 희생인 셈이다. 면역학과 줄기세포를 연구한 의학도에서 해부학자로 의대생들을 교육함과 더불어, 이런 연구를 할 수 있는 연구자라는 것이 일생일대의 가장 큰 행운이라고 생각하고 재미있게 연구를 진행하고 있는 중이다. 지면의 부족함으로 실험적 방법론과 분석 방향에 대해서 자세하게 서술하지 못한 점이 아쉬운 것이 사실이나, 새로운 관점으로 해부학 연구를 진행하고 싶은 연구자들은 언제나 환영이다.
1. Wilson, E.B. (1892). The Cell-lineage of Nereis. A contribution to the cytogeny of the Annelid body. J. Morphol. VI, 368.
2. Conklin, E.G. (1905). The organization and cell lineage of the ascidian egg. J. Acad. Nat. Sci. Phila. 13, 1–119.
3. Sulston, J. E., & Horvitz, H. R. (1977). Post-embryonic cell lineages of the nematode, Caenorhabditis elegans. Developmental biology, 56(1), 110-156.
4. Sulston, J.E., Schierenberg, E., White, J.G., and Thomson, J.N. (1983). The embryonic cell lineage of the nematode Caenorhabditis elegans. Dev. Biol. 100, 64–119.
5. Harrison DA, Perrimon N: Simple and efficient generation of marked clones in Drosophila. Curr Biol 1993, 3:424-433.
6. McKenna, A., Findlay, G. M., Gagnon, J. A., Horwitz, M. S., Schier, A. F., & Shendure, J. (2016). Whole-organism lineage tracing by combinatorial and cumulative genome editing. Science, 353(6298), aaf7907.
7. Spanjaard, B., Hu, B., Mitic, N., Olivares-Chauvet, P., Janjuha, S., Ninov, N., & Junker, J. P. (2018). Simultaneous lineage tracing and cell-type identification using CRISPR–Cas9-induced genetic scars. Nature biotechnology, 36(5), 469.
8. Kalhor, R., Kalhor, K., Mejia, L., Leeper, K., Graveline, A., Mali, P., & Church, G. M. (2018). Developmental barcoding of whole mouse via homing CRISPR. Science, 361(6405), eaat9804.
9. Livet, J., Weissman, T. A., Kang, H., Draft, R. W., Lu, J., Bennis, R. A., ... & Lichtman, J. W. (2007). Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system. Nature, 450(7166), 56.
10. Snippert, H. J., Van Der Flier, L. G., Sato, T., Van Es, J. H., Van Den Born, M., Kroon-Veenboer, C., ... & Clevers, H. (2010). Intestinal crypt homeostasis results from neutral competition between symmetrically dividing Lgr5 stem cells. Cell, 143(1), 134-144.
11. Vogelstein, B., Papadopoulos, N., Velculescu, V. E., Zhou, S., Diaz, L. A., & Kinzler, K. W. (2013). Cancer genome landscapes. science, 339(6127), 1546-1558.
12. James, C. D., Carlbom, E., Nordenskjold, M., Collins, V. P., & Cavenee, W. K. (1989). Mitotic recombination of chromosome 17 in astrocytomas. Proceedings of the National Academy of Sciences, 86(8), 2858-2862.
13. McFarland, C. D., Yaglom, J. A., Wojtkowiak, J. W., Scott, J. G., Morse, D. L., Sherman, M. Y., & Mirny, L. A. (2017). The damaging effect of passenger mutations on cancer progression. Cancer research.
14. Gerlinger, M., Rowan, A. J., Horswell, S., Larkin, J., Endesfelder, D., Gronroos, E., ... & Varela, I. (2012). Intratumor heterogeneity and branched evolution revealed by multiregion sequencing. New England journal of medicine, 366(10), 883-892.
15. Kreso, A., & Dick, J. E. (2014). Evolution of the cancer stem cell model. Cell stem cell, 14(3), 275-291.
16. Landau, D. A., Carter, S. L., Stojanov, P., McKenna, A., Stevenson, K., Lawrence, M. S., ... & Wan, Y. (2013). Evolution and impact of subclonal mutations in chronic lymphocytic leukemia. Cell, 152(4), 714-726.
17. Sottoriva, A., Spiteri, I., Piccirillo, S. G., Touloumis, A., Collins, V. P., Marioni, J. C., ... & Tavaré, S. (2013). Intratumor heterogeneity in human glioblastoma reflects cancer evolutionary dynamics. Proceedings of the National Academy of Sciences, 110(10), 4009-4014.
18. Behjati, S., Huch, M., van Boxtel, R., Karthaus, W., Wedge, D. C., Tamuri, A. U., ... & Tarpey, P. S. (2014). Genome sequencing of normal cells reveals developmental lineages and mutational processes. Nature, 513(7518), 422.
19. Ju, Y. S., Martincorena, I., Gerstung, M., Petljak, M., Alexandrov, L. B., Rahbari, R., ... & Martin, S. (2017). Somatic mutations reveal asymmetric cellular dynamics in the early human embryo. Nature, 543(7647), 714.
20. Bae, T., Tomasini, L., Mariani, J., Zhou, B., Roychowdhury, T., Franjic, D., ... & Riley-Gillis, B. (2018). Different mutational rates and mechanisms in human cells at pregastrulation and neurogenesis. Science, 359(6375), 550-555.
21. Lodato, M. A., Rodin, R. E., Bohrson, C. L., Coulter, M. E., Barton, A. R., Kwon, M., ... & Yang, P. (2018). Aging and neurodegeneration are associated with increased mutations in single human neurons. Science, 359(6375), 555-559.
첨부파일